1. ParaAT
参考blog.ParaAT:https://yanzhongsino.github.io/2021/10/29/bioinfo_align_pep2cds/
+2. KaKs_Calculator 2.0
KaKs_Calculator 2.0工具包包含了17种计算Ka和Ks的方法,包含Gamma系列,并可识别基于蛋白编码序列的滑动窗口,基于C++和Java,在Windows和Linux平台都可使用。
+3. Ka和Ks计算
-
+
- ParaAT比对指定的基因对的氨基酸序列,并转化成比对的CDS序列,并可指定输出格式,如axt格式。 +
- KaKs_Calculator用于计算比对好的基因对的kaks。 +
用还会用到两个脚本:
+-
+
- axt2one-line.py:https://github.com/scbgfengchao/4DTv/blob/master/axt2one-line.py转换axt格式为单行 +
- calculate_4DTV_correction.pl:https://github.com/JinfengChen/Scripts/blob/master/FFgenome/03.evolution/distance_kaks_4dtv/bin/calculate_4DTV_correction.pl计算4dtv。 +
4. 软件准备
-
+
- 安装ParaAT.pl +
-
+
- 参考blog.ParaAT:https://yanzhongsino.github.io/2021/10/29/bioinfo_align_pep2cds/ +
wget ftp://download.big.ac.cn/bigd/tools/ParaAT2.0.tar.gz |
-
+
- 安装KaKs_Calculator2.0
KaKs_Calculator2.0下载地址:https://sourceforge.net/projects/kakscalculator2/?source=typ_redirect
+
wget https://altushost-swe.dl.sourceforge.net/project/kakscalculator2/KaKs_Calculator2.0.tar.gz |
-
+
- 然后把
./KaKs_Calculator2.0/bin/Linux/和./KaKs_Calculator2.0/src/添加到环境变量即可使用KaKs_Calculator和AXTConvertor命令。
+
5. 文件准备
ParaAT.pl需要三个输入文件,参考blog.ParaAT:https://yanzhongsino.github.io/2021/10/29/bioinfo_align_pep2cds/
+-
+
- sample.id +
-
+
- 两列,每行对应两条要做成对比对的序列ID; +
- 任意行,ParaAT可以批量处理多个成对比对。 +
- 一个例子:
cat sample.collinearity |grep "species_prefix"|cut -f2,3 >sample.id用MCScanX的结果文件提取blocks的同源gene对,获得sample.id文件。
+
-
+
- cds.fa +
-
+
- 包括所有需要比对的cds序列的文件 +
-
+
- pep.fa +
-
+
- 包括所有需要比对的蛋白序列的文件 +
6. 操作步骤
6.1. 用ParaAT获取基因对比对序列
ParaAT比对sample.id指定的基因对的氨基酸序列,并转化成比对的CDS序列,并可指定输出为axt格式。
+-
+
- 命令 +
echo "24" >proc #指定ParaAT.pl使用线程 |
-
+
- notes +
-
+
- axt格式包括三行,第一行两个序列ID之间用短横杠-相连,第二行第一条序列,第三行第二条序列。 +
- 建议加上-g和-t,免得后面计算Ks时报错
Error. The size of two sequences in 'ctg00816-ctg08844' is not equal。
+ - ParaAT.pl命令中加上-k参数可以在获得axt文件后自动调用KaKs_Calculator计算kaks值,使用MA模型,比YN模型慢很多,推荐不加-k参数,而是手动用KaKs_Calculator的YN模型,生成sample.axt_yn.kaks文件。 +
6.2. 用KaKs_Calculator计算基因对的Ka、Ks和4dtv值
ParaAT.pl的-k参数只能指定KaKs_Calculator的MA模型计算kaks值,如果需要指定其他的模型,则可以手动运行计算。
+KaKs_Calculator可计算比对好的CDS序列的Ka和Ks。
+-
+
- 计算Ka和Ks +
-
+
- 获得all.kaks文件
cd sample.paraat # 进入ParaAT.pl生成的文件夹
for i in `ls *axt`;do KaKs_Calculator -i $i -o ${i}_yn.kaks -m YN;done #用YN模型计算每个gene对的KaKs,生成四列数据,gene对,Ka,Ks,Ka/Ks
for i in `ls |grep "_yn.kaks"`;do awk 'NR>1{print $1"\t"$3"\t"$4"\t"$5}' $i >>../all.kaks;done #合并kaks到all.kaks文件
+
-
+
- 计算4dtv +
-
+
- 获得all.4dtv文件
cd sample.paraat # 进入ParaAT.pl生成的文件夹
for i in `ls *axt`;do axt2one-line.py $i ${i}.one-line;done #多行axt文件转换成单行
ls |grep "axt.one-line"|while read id;do calculate_4DTV_correction.pl $id > ${id%%one-line}4dtv;done #计算4dtv值,生成两列数据,gene对,4dtv
for i in `ls |grep "4dtv"`;do awk 'NR>1{print $1"\t"$3}' $i >>../all.4dtv;done #合并4dtv值到all.4dtv
+
-
+
- 合并和整理结果 +
-
+
- 获得all.results文件
cd .. #返回上一级目录
join all.kaks all.4dtv |sed "s/ /\t/g" |awk '$3 != "NA" {print $0}' |sed '1i\genepair\tKa\tKs\tKa/Ks\t4dtv_corrected' >all.results #以gene对为基准,join合并kaks和4dtv值到一个文件,然后过滤Ks值为NA的无效数据,添加标题行。
rm all.kaks* all.4dtv* #删除中间文件
+
7. references
-
+
- ParaAT paper:https://www.sciencedirect.com/science/article/pii/S0006291X12003518 +
- KaKs_Calculator2.0 github:https://github.com/kullrich/kakscalculator2 +
- KaKs_Calculator2.0 paper:https://www.sciencedirect.com/science/article/pii/S1672022910600083?via%3Dihub +
+
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
+
]]>
+ (全文约4000字)
-clusterProfiler相关的博客共有三篇,共同食用,效果更好 :wink: :
--
-
- 博客富集分析:(三)clusterProfiler概述 -
- 博客富集分析:(四) clusterProfiler:不同物种的GO+KEGG富集分析 -
- 博客富集分析:(五)clusterProfiler:Visualization -
1. 可视化的输入数据
clusterProfiler的可视化一般只支持clusterProfiler富集分析结果的可视化,通过认识clusterProfiler可视化接受的输入数据的格式,可以修改其他富集分析结果文件的格式,来用clusterProfiler进行可视化绘图。
-1.1. 可视化输入数据格式
-
-
- 查看ego格式
clusterProfiler的可视化包接受的输入数据是前面富集分析得到的结果(比如ego/kk),用str(ego)或class(ego)可以看到ego的格式是叫enrichResult的R的数据类型。library(clusterProfiler)
> class(ego) #查看ego的数据类型/类
[1] "enrichResult"
attr(,"package")
[1] "DOSE"
-
如果手头没有ego数据,可以用clusterProfiler的样例数据快速得到一个edo,与ego格式一样。
-library(clusterProfiler) |
-
-
enrichResult(R的class类型)格式
-
在DOSE包中查到,enrichResult具体格式如下:+setClass("enrichResult",
representation=representation(
result = "data.frame",
pvalueCutoff = "numeric",
pAdjustMethod = "character",
qvalueCutoff = "numeric",
organism = "character",
style="margin: 0px; padding: 0px; color: rgb(221, 17, 68);">"character",
gene = "character",
keytype = "character",
universe = "character",
gene2Symbol = "character",
geneSets = "list",
readable = "logical"
),
prototype=prototype(readable = FALSE)
)(全文约6600字)
+1. 富集分析
1.1. 富集分析概念
-
+
富集分析
富集分析,本质上是对数据的分布检验,如果分布集中在某个区域,则认为富集。
常用的分布检验方法有卡方检验、Fisher精确检验以及KS检验等方法。
-result变量格式
-
enrichResult中最重要的是result,是储存富集结果的dataframe。
result变量与clusterProfiler富集分析中保存ego的结果文件是一致的。
-
- -ego@result[c(13,14),] #查看ego的result变量的13,14行
ONTOLOGY ID Description GeneRatio BgRatio pvalue p.adjust qvalue geneID Count
13 BP GO:0010051 xylem and phloem pattern formation 3/349 129/16975 1.431350e-05 0.001294821 0.001099880 mc40782/mc40784/mc40918 3
14 BP GO:0048598 embryonic morphogenesis 2/349 131/16975 1.673394e-05 0.001405651 0.001194023 mc40784/mc40918 2一般而言result有9列。这里因为用enrichGO富集时ont参数选择ALL,结果就会在第一列前多一列ONTOLOGY。
--
-
- 第一列是ID,也就是富集通路的编号(GO:0010222); -
- 第二列是Description,也就是富集通路的名称; -
- 第三列是GeneRatio,也就是要富集的基因中在对应通路中的比例; -
- 第4列是BgRation,也就是对应通过的基因在全基因组注释中的比例; -
- 第5,6,7列都是统计检验的结果; -
- 第8列是geneID,也就是富集到基因的名字,多个geneID是以斜线隔开的; -
- 第9列是Count,也就是富集到的基因数目。 -
1.2. 输入数据准备
根据不同情况为clusterProfiler的可视化准备输入数据。
--
-
接着clusterProfiler富集分析做可视化
-
如果是接着clusterProfiler的enrichGO(),gseGO(),enricher(),gseGO()等函数的结果ego,不要关闭R环境,在R里直接进行用于下一步可视化即可。
-保存的clusterProfiler富集分析结果做可视化
-
-
-
-
- 如果是clusterProfiler的enrichGO(),gseGO(),enricher(),gseGO()等函数的结果
ego保存成的文件,已关闭R环境。
- - 可导入文件,新建enrichResult对象ego,再进行下一步可视化。 -
- 这里假设用R命令
write.table(as.data.frame(ego),"go_enrich.csv",sep="\t",row.names =F,quote=F)保存ego在go_enrich.csv文件。data<-read.table("go_enrich.csv",sep="\t",header=T,quote="")
head(data,2) #查看data前2行
ONTOLOGY ID Description GeneRatio
1 BP GO:0010222 stem vascular tissue pattern formation 12/349
2 BP GO:0010588 cotyledon vascular tissue pattern formation 12/349
BgRatio pvalue p.adjust qvalue
1 29/16975 1.792157e-13 2.107577e-10 1.790270e-10
2 39/16975 1.122611e-11 6.600951e-09 5.607145e-09
geneID
1 mc11300/mc11301/mc19080/mc19081/mc26300/mc31693/mc37850/mc40780/mc40781/mc40782/mc40784/mc40918
2 mc11300/mc11301/mc19080/mc19081/mc26300/mc31693/mc37850/mc40780/mc40781/mc40782/mc40784/mc40918
Count
1 12
2 12
geneID_all <- unlist(apply(as.matrix(data$geneID),1,function(x) unlist(strsplit(x,'/')))) #得到富集到的所用geneID
ego<-new("enrichResult", result=data, gene=geneID_all, pvalueCutoff=0.01,pAdjustMethod="BH",qvalueCutoff=0.05,ontology="BP",keytype="GID",universe='Unknown',geneSets=list(),organism="Unknown",readable=FALSE) #把data内容赋值给ego的result,geneID_all赋值给gene,每个富集到的GO对应的gene集应该赋值给geneSets(数据是字典(键值对是GOID和geneIDs)组成的列表,这里直接给了空的),ontology与enrichGO分析的ont参数一致,这里的pvalueCutoff=0.01,pAdjustMethod="BH",qvalueCutoff=0.05根据富集分析参数的设置,或者随意设置或者不设置也不会影响可视化。
-
-
-
- 其他来源富集分析结果可视化
如果是其他软件的富集分析结果,可以根据ego的result变量格式进行修改格式,改成go_enrich.csv相同的格式的文件,再按照上面的步骤导入文件,并保存到新建的ego对象。即可用clusterProfiler的可视化包可视化其他软件的富集分析结果了。
-
2. 功能富集结果可视化
下面的可视化大多基于在R中已获得富集分析的结果ego。
-2.1. enrichplot包
enrichplot包有几种可视化方法来解释富集结果,支持clusterProfiler获得的ORA和GSEA富集结果。
-2.1.1. 安装和载入
安装和载入enrichplot包
-- -BiocManager::install("enrichplot")
library(enrichplot)2.1.2. 可视化包
-
-
- 推荐dotplot或barplot可视化前10个GO Terms条目。 -
- 推荐goplot有向无环图查看富集的GO Terms间的关系。 -
-
-
- 可视化barplot —— 条形图
将富集分数(例如p 值)和基因计数或比率描述为条形高度和颜色。横轴为该GO term下的差异基因个数,纵轴为富集到的GO Terms的描述信息, showCategory指定展示的GO Terms的个数为20个,默认展示显著富集的top10个,即p.adjust最小的10个。
-
-barplot(ego, showCategory=20, title="EnrichmentGO_MF")使用mutate导出的其他变量也可以用作条形高度或颜色。
-- -mutate(ego, qscore = -log(p.adjust, base=10)) %>%
barplot(x="qscore")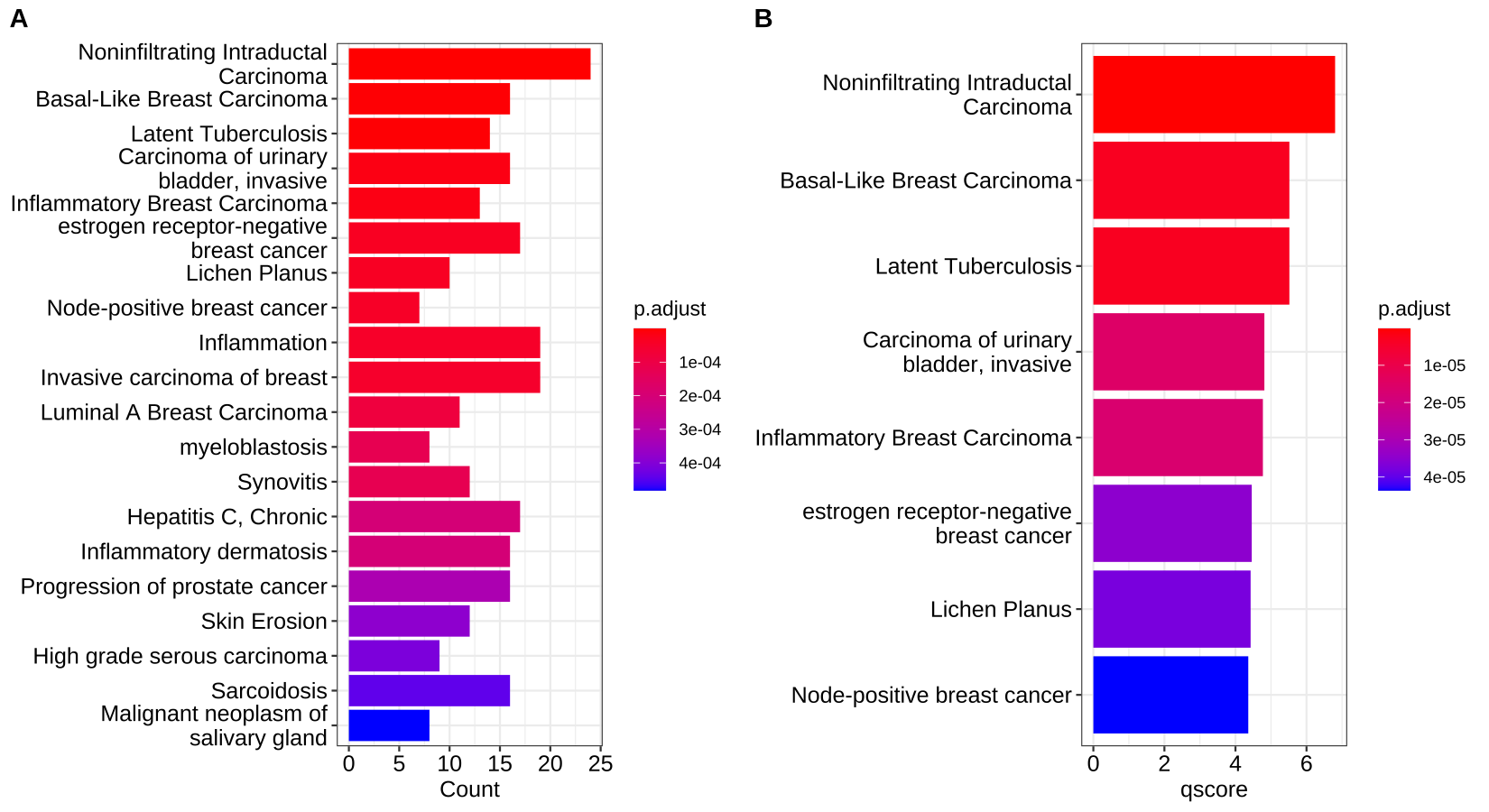 -
-
-
-
-Figure 1. Bar plot of enriched terms
from clusterProfiler book-
-
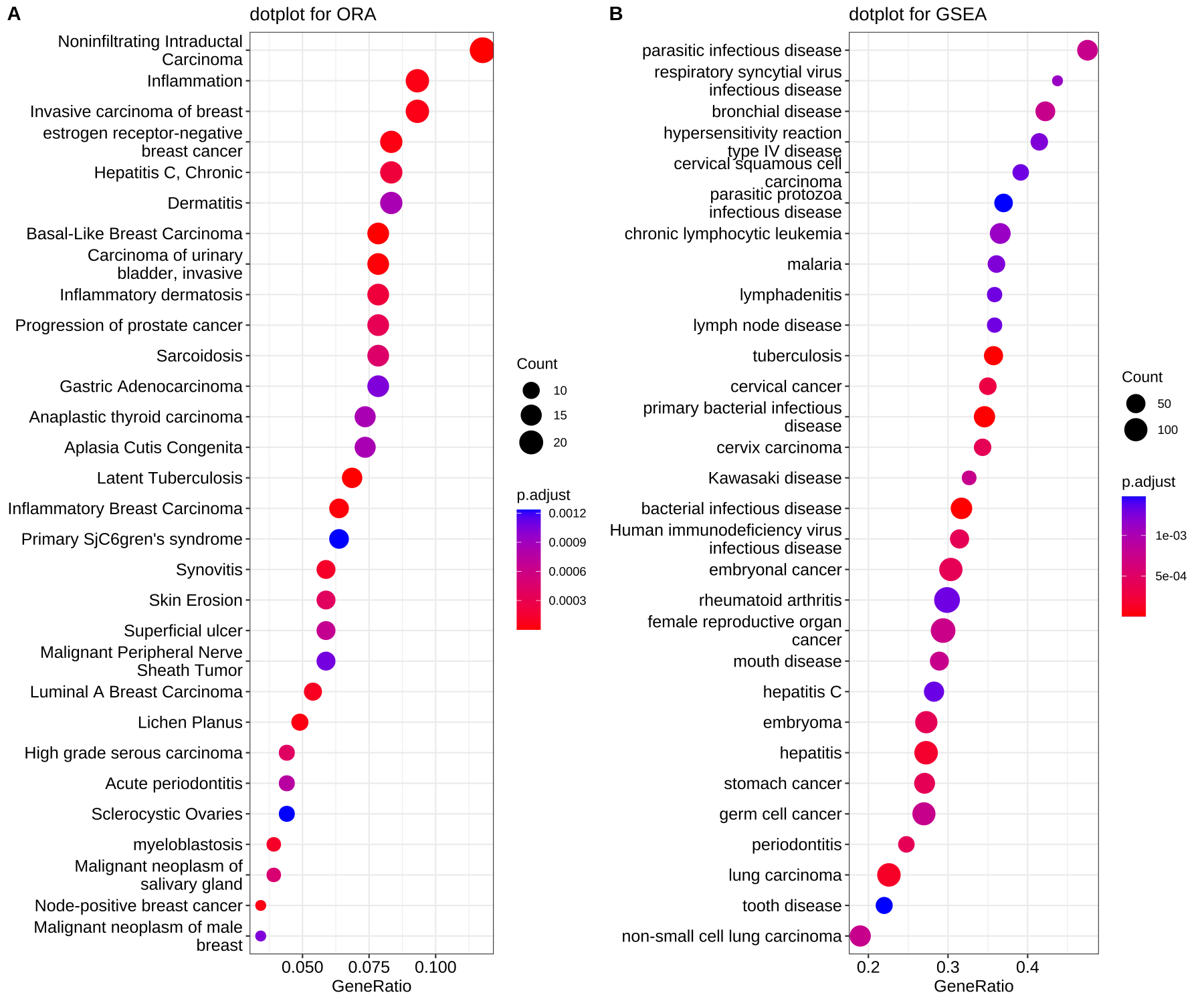
- 可视化dotplot —— 点阵图
dotplot(edo, showCategory=30) + ggtitle("dotplot for ORA")
-
-dotplot(edo2, showCategory=30) + ggtitle("dotplot for GSEA")散点图,横坐标为GeneRatio,纵坐标为富集到的GO Terms的描述信息,showCategory指定展示的GO Terms的个数,默认展示显著富集的top10个,即p.adjust最小的10个。
- -
-
-
-
-Figure 2. Dot plot of enriched terms
from clusterProfiler book-
-
- 可视化cnetplot —— 类别网络图
cnetplot 将基因和生物学概念(例如 GO 术语或 KEGG 通路)的联系描述为一个网络(有助于查看哪些基因涉及富集通路和可能属于多个注释类别的基因)。对于基因和富集的GO terms之间的对应关系进行展示,如果一个基因位于一个GO Terms下,则将该基因与GO连线。图中灰色的点代表基因,黄色的点代表富集到的GO terms, 默认画top5富集到的GO terms, GO 节点的大小对应富集到的基因个数。
-
-cnetplot(ego, categorySize = "pvalue", foldChange = gene_list- -## convert gene ID to Symbol
edox <- setReadable(ego, 'org.Hs.eg.db', 'ENTREZID')
p1 <- cnetplot(edox, foldChange=geneList)
## categorySize can be scaled by 'pvalue' or 'geneNum'
p2 <- cnetplot(edox, categorySize="pvalue", foldChange=geneList)
p3 <- cnetplot(edox, foldChange=geneList, circular = TRUE, colorEdge = TRUE)
cowplot::plot_grid(p1, p2, p3, ncol=3, labels=LETTERS[1:3], rel_widths=c(.8, .8, 1.2))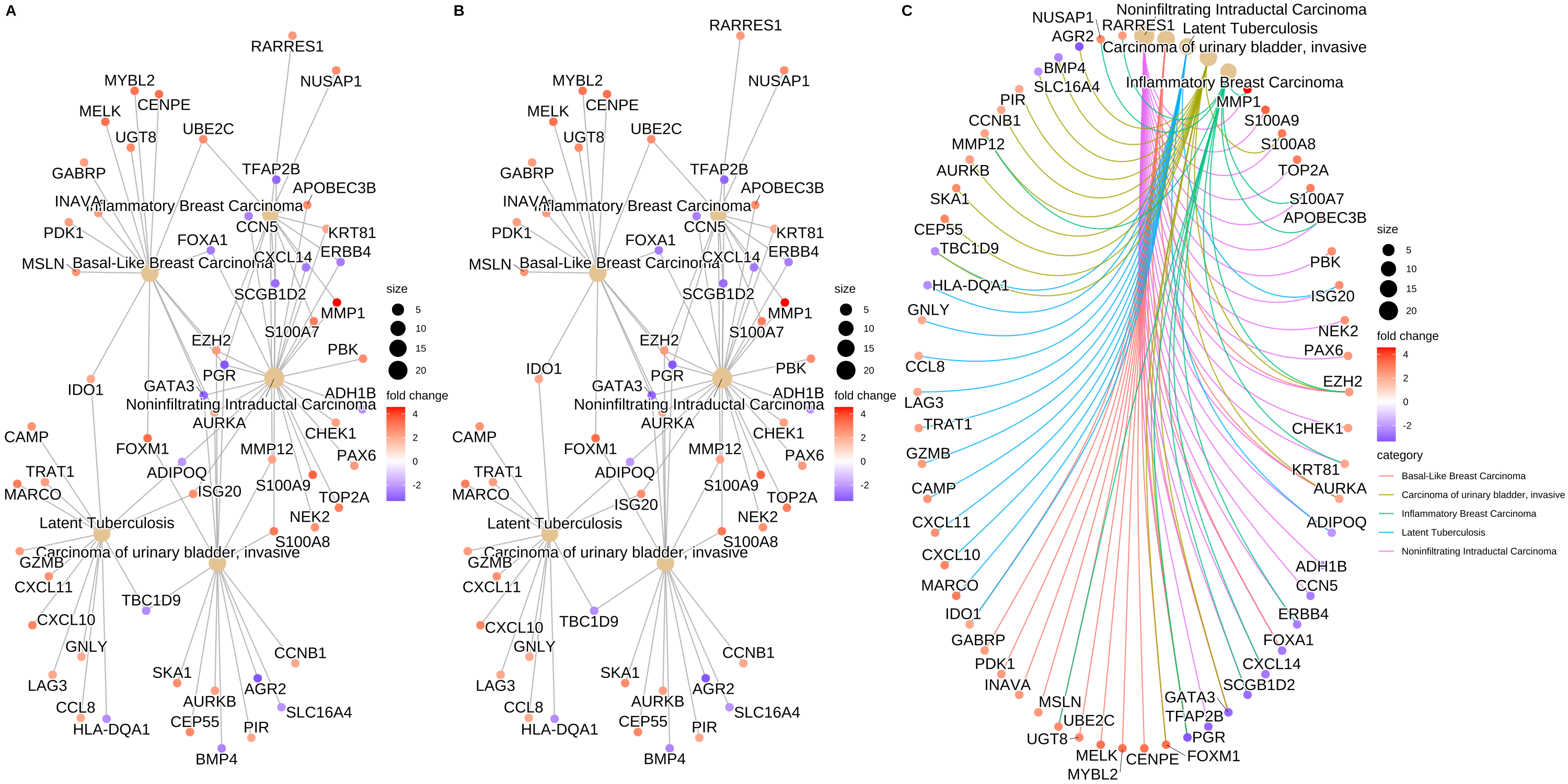 -
-
-
-
-Figure 3. Network plot of enriched terms
from clusterProfiler book-
-
- 可视化heatplot —— 类热图功能分类
同样使用edox。
heatplot类似cnetplot,而显示为热图的关系。
如果用户想要显示大量重要术语,那么类别网络图可能会过于复杂。在heatplot能够简化结果和更容易识别的表达模式。
-
- -p1 <- heatplot(edox, showCategory=5)
p2 <- heatplot(edox, foldChange=geneList, showCategory=5)
cowplot::plot_grid(p1, p2, ncol=1, labels=LETTERS[1:2])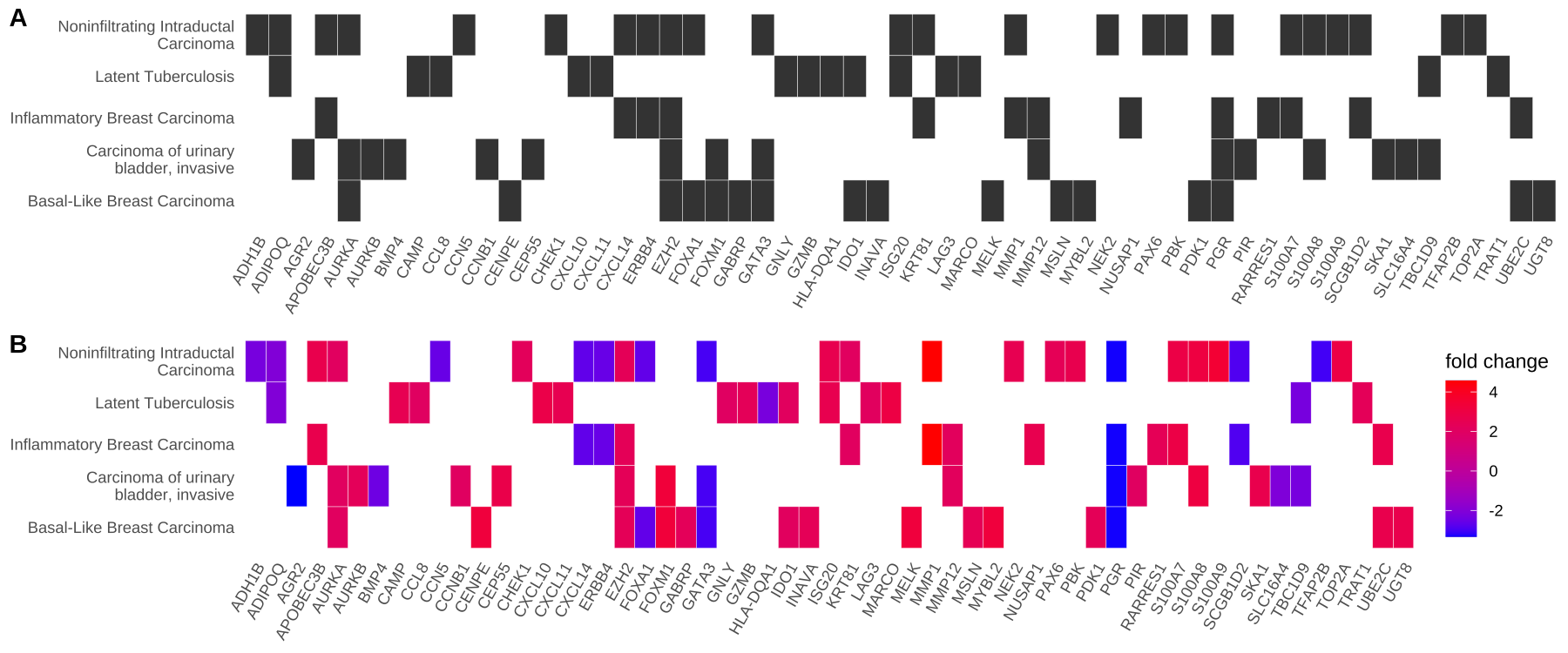 -
-
-
-
-Figure 4. Heatmap plot of enriched terms
from clusterProfiler book-
-
- 可视化treeplot —— 树状图
treeplot()函数执行丰富术语的层次聚类。它依赖于pairwise_termsim()函数计算的丰富项的成对相似性,默认情况下使用 Jaccard 的相似性指数 (JC)。如果支持,用户还可以使用语义相似度值(例如,GO、DO和MeSH)。
-
默认聚合方法treeplot()是ward.D,用户可以通过hclust_method参数指定其他方法(例如,’average’、’complete’、’median’、’centroid’等。
-treeplot()函数会将树切割成几个子树(由nCluster参数指定(默认为 5))并使用高频词标记子树。
-- -edox2 <- pairwise_termsim(edox)
p1 <- treeplot(edox2)
p2 <- treeplot(edox2, hclust_method = "average")
aplot::plot_list(p1, p2, tag_levels='A')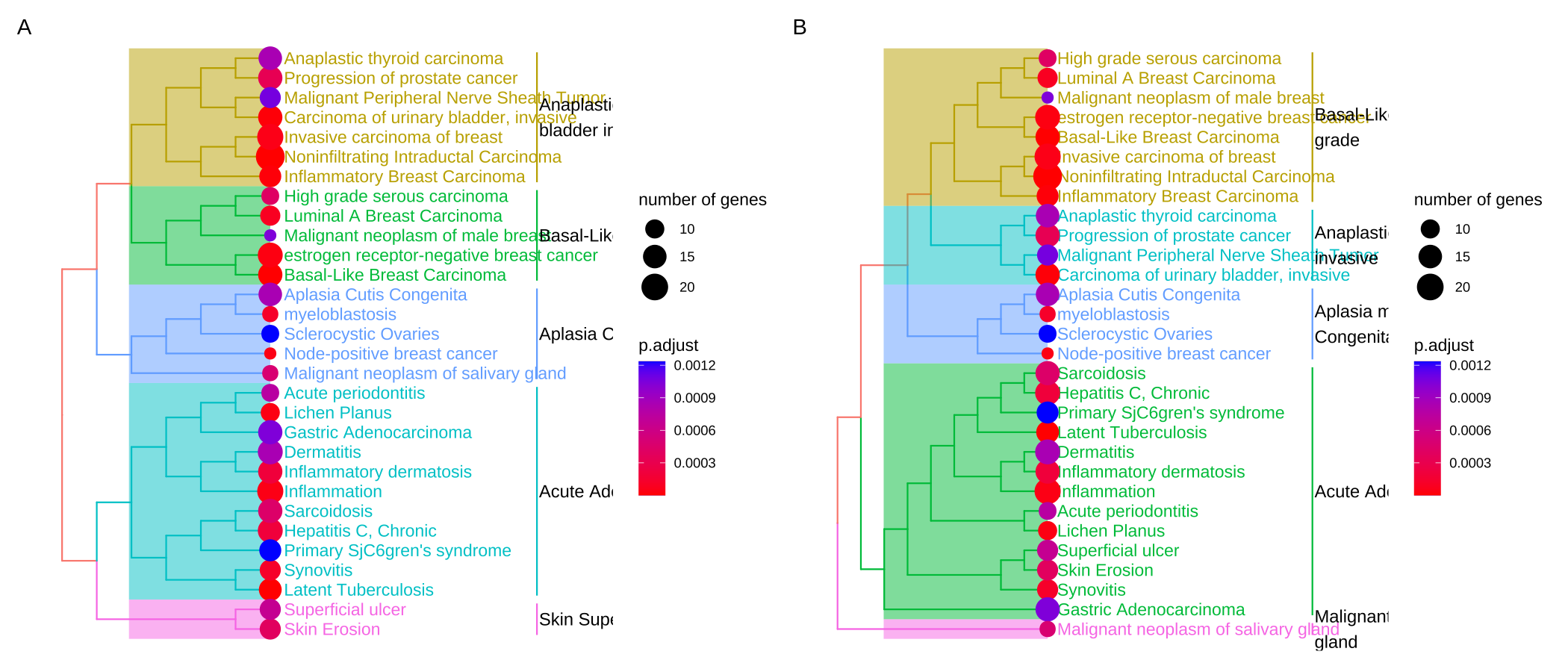 -
-
-
-
-Figure 5. Tree plot of enriched terms
from clusterProfiler book-
-
- 可视化emapplot —— 富集图
对于富集到的GO terms之间的基因重叠关系进行展示,如果两个GO terms系的差异基因存在重叠,说明这两个节点存在overlap关系,在图中用线条连接起来。每个节点是一个富集到的GO term, 默认画top30个富集到的GO terms, 节点大小对应该GO terms下富集到的差异基因个数,节点的颜色对应p.adjust的值,从小到大,对应蓝色到红色。
-
- -ego2 <- pairwise_termsim(ego)
p1 <- emapplot(ego2)
p2 <- emapplot(ego2, cex_category=1.5)
p3 <- emapplot(ego2, layout="kk")
p4 <- emapplot(ego2, cex_category=1.5,layout="kk")
cowplot::plot_grid(p1, p2, p3, p4, ncol=2, labels=LETTERS[1:4])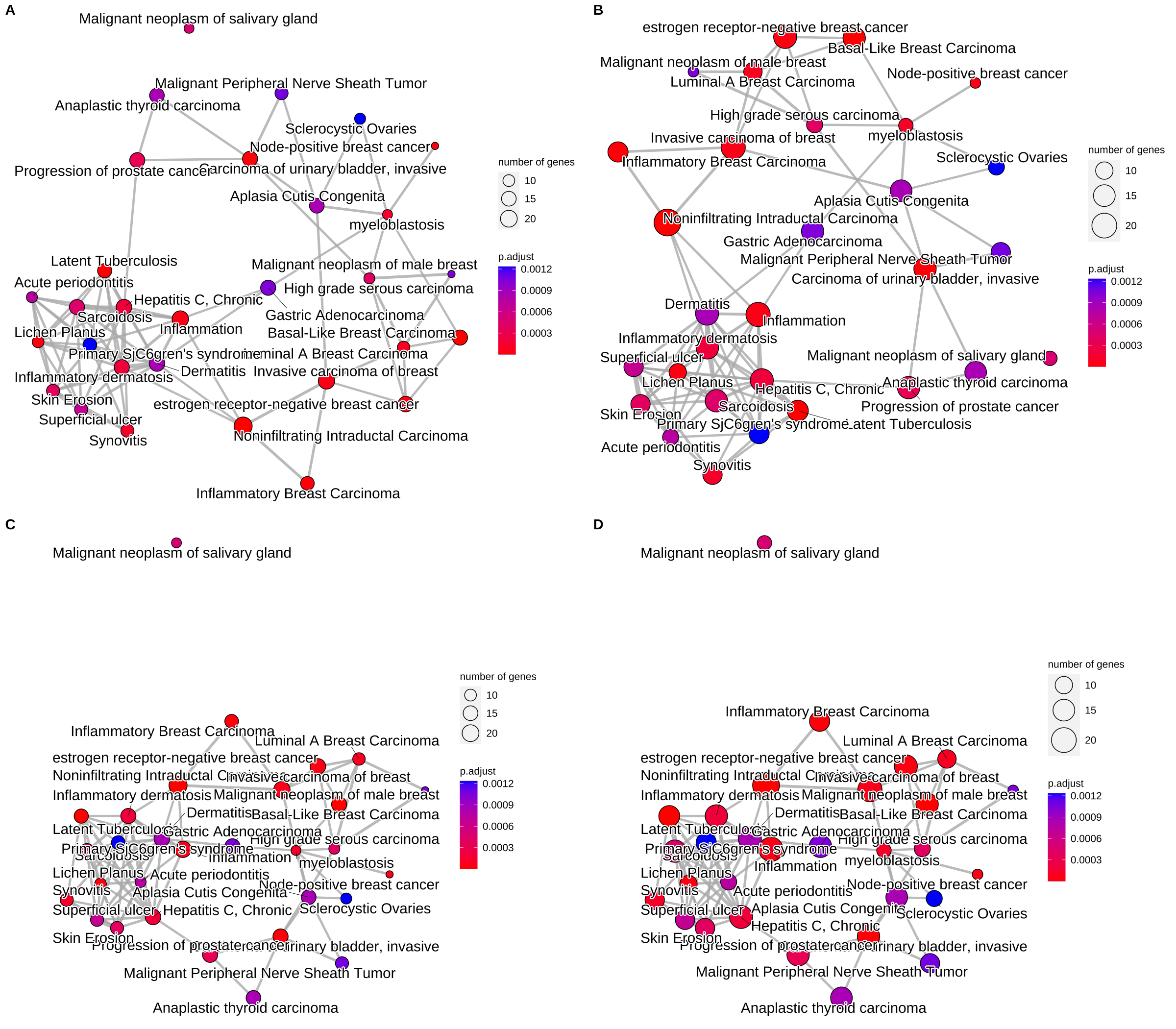 -
-
-
-
-Figure 6. Plot for results obtained from hypergeometric test and gene set enrichment analysis. default (A), cex_category=1.5 (B), layout=”kk” (C) and cex_category=1.5,layout=”kk” (D).
from clusterProfiler book-
-
- 可视化upsetplot —— upset图
upsetplot是cnetplot可视化基因和基因集之间复杂关联的替代方法。它强调不同基因集之间的基因重叠。
-
-upsetplot(ego)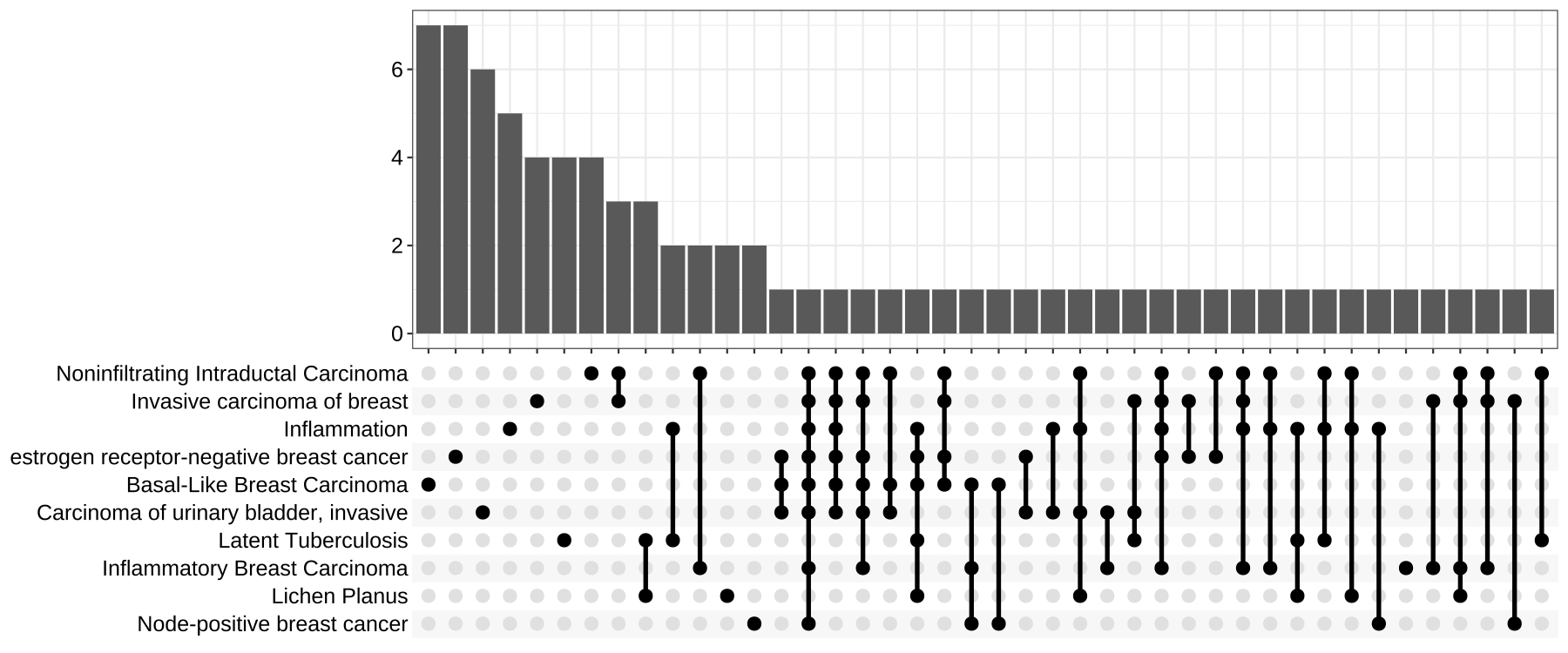 -
-
-
-
-Figure 7. Upsetplot for over-representation analysis.
from clusterProfiler book-
-
- 可视化ridgeplot —— 脊线图
ridgeplot将可视化核心富集基因的表达分布为GSEA富集类别。它帮助用户解释上调/下调的途径。
-
-ridgeplot(ego)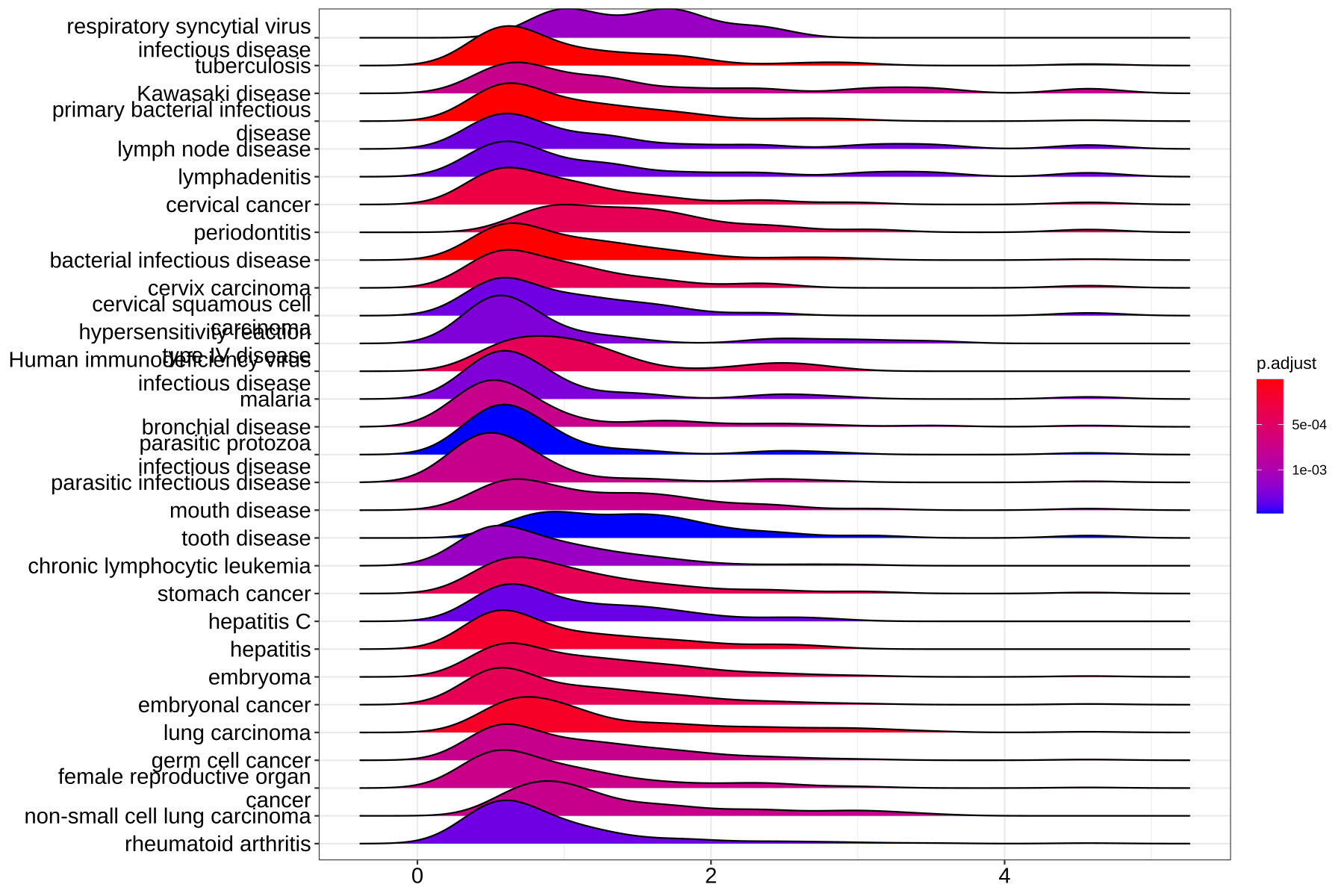 -
-
-
-
-Figure 8. Ridgeplot for gene set enrichment analysis.
from clusterProfiler book2.2. 可视化plotGOgraph/goplot —— 有向无环图
-
-
plotGOgraph(ego, firstSigNodes=10)
-
-
-
- 有向无环图(Directed acyclic graph, DAG),矩形代表富集到的top10个GO Terms,颜色从黄到红,对应p值从大到小。和topGO做富集分析的DAG图一样。 -
当enrichGO富集分析时ont参数选了ALL时,结果文件会在第一列前增加一列ONTOLOGY为子类,这时直接用于plotGOgraph画图会报错。
-
试了下,下面两种方案还是会报错Error in if (!ont %in% c(“BP”, “MF”, “CC”)) { :argument is of length zero。。还是尽量在enrichGO分析时就用ont=”BP”吧。-
-
- 可以在结果文件中筛选出特定子类(比如BP)的结果行,并删除第一列ONTOLOGY后保存文件,再读进R用于plotGOgraph画图。 -
- 也可以在R内用命令
ego2<-ego%>%filter(ONTOLOGY== "BP")筛选BP子类,接着用ego3<-ego2%>%select(!ONTOLOGY)或者ego3<-ego2[,-1]删除第一列(即ONTOLOGY列),然后用plotGOgraph(ego3)作图。
-
 -
-
-
-
-Figure 9. DAG图
from clusterProfiler blog-
-
goplot(ego, showCategory = 10)
-
-
-
- igraph布局方式的有向无环图 -
 -
-
-
-
-Figure 10. goplot的DAG图
from clusterProfiler book2.3. 可视化 —— wordcloud
词云的方式显示结果
-- -install.packages("wordcloud")
library(wordcloud)
wcdf <- read.table(text = ego$GeneRatio, sep = "/")[1]
wcdf$term <- ego[,2]
wordcloud(words = wcdf$term, freq = wcdf$V1, scale=(c(4, .1)), colors=brewer.pal(8, "Dark2"), max.words = 25) -
-
-
-
-Figure 11. wordcloud词云图
from NGS Analysis ebook3. 导出可视化结果
-
-
- Rstudio -
如果是在Rstudio中,可以直接看到绘图结果,导出需要的文件格式即可。
--
-
- 代码导出
pdf("ego.pdf") #如果保存png,就改成png("ego.png")
ego_fig<-barplot(x) #画图函数
print(ego_fig) #画到pdf文件
dev.off() #关闭pdf画板
-
4. references
-
-
- clusterProfiler github:https://github.com/YuLab-SMU/clusterProfiler -
- clusterProfiler paper:https://www.cell.com/the-innovation/fulltext/S2666-6758(21)00066-7?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS2666675821000667%3Fshowall%3Dtrue -
- clusterProfiler book:http://yulab-smu.top/biomedical-knowledge-mining-book/index.html -
- clusterProfiler manual:https://bioconductor.org/packages/devel/bioc/manuals/clusterProfiler/man/clusterProfiler.pdf -
- clusterProfiler ducumentation:https://guangchuangyu.github.io/software/clusterProfiler/documentation/ -
- 其他来源结果可视化:https://cloud.tencent.com/developer/article/1613815 -
- wordcloud:https://learn.gencore.bio.nyu.edu/rna-seq-analysis/over-representation-analysis/ -
--
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 -]]>
-]]>
(全文约6600字)
-1. 富集分析
1.1. 富集分析概念
-
-
富集分析
-
富集分析,本质上是对数据的分布检验,如果分布集中在某个区域,则认为富集。
常用的分布检验方法有卡方检验、Fisher精确检验以及KS检验等方法。
-生物信息学领域的富集分析
+
在 背景基因集(N) 下获得 一组特定基因集(S) ,S可能是基因列表,表达图谱,基因芯片等形式。在预先构建好基因注释数据库(例如GO,KEGG等)已对背景基因集(N)根据生物功能或过程进行分类的前提下,通过统计学算法找出有那些显著区别于背景基因集(N)的类别(生物组成/功能/过程),或者找出这组特定基因集间在生物组成/功能/过程的共性,经过聚类后去除冗余得到基因富集结果的过程,即为富集分析。生物信息学领域的富集分析
在 背景基因集(N) 下获得 一组特定基因集(S) ,S可能是基因列表,表达图谱,基因芯片等形式。在预先构建好基因注释数据库(例如GO,KEGG等)已对背景基因集(N)根据生物功能或过程进行分类的前提下,通过统计学算法找出有那些显著区别于背景基因集(N)的类别(生物组成/功能/过程),或者找出这组特定基因集间在生物组成/功能/过程的共性,经过聚类后去除冗余得到基因富集结果的过程,即为富集分析。
可以这样简单理解富集分析在做什么。全国人口的户籍作为背景数据,我们通过富集分析可以知道相对于全国背景,客家人是不是明显在广东聚集。比如如果广东的客家人数/全国客家人数这个比值远超过广东人数/全国人数的比值,那么我们可以说客家人在广东是富集的。
@@ -2427,15 +2232,212 @@ BiocManager的版本与R版本一一对应,安装时如果版本不对会有+
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>(全文约4000字)
+clusterProfiler相关的博客共有三篇,共同食用,效果更好 :wink: :
+-
+
- 博客富集分析:(三)clusterProfiler概述 +
- 博客富集分析:(四) clusterProfiler:不同物种的GO+KEGG富集分析 +
- 博客富集分析:(五)clusterProfiler:Visualization +
1. 可视化的输入数据
clusterProfiler的可视化一般只支持clusterProfiler富集分析结果的可视化,通过认识clusterProfiler可视化接受的输入数据的格式,可以修改其他富集分析结果文件的格式,来用clusterProfiler进行可视化绘图。
+1.1. 可视化输入数据格式
-
+
- 查看ego格式
clusterProfiler的可视化包接受的输入数据是前面富集分析得到的结果(比如ego/kk),用str(ego)或class(ego)可以看到ego的格式是叫enrichResult的R的数据类型。library(clusterProfiler)
> class(ego) #查看ego的数据类型/类
[1] "enrichResult"
attr(,"package")
[1] "DOSE"
+
如果手头没有ego数据,可以用clusterProfiler的样例数据快速得到一个edo,与ego格式一样。
+library(clusterProfiler) |
-
+
enrichResult(R的class类型)格式
+
在DOSE包中查到,enrichResult具体格式如下:+setClass("enrichResult",
representation=representation(
result = "data.frame",
pvalueCutoff = "numeric",
pAdjustMethod = "character",
qvalueCutoff = "numeric",
organism = "character",
style="margin: 0px; padding: 0px; color: rgb(221, 17, 68);">"character",
gene = "character",
keytype = "character",
universe = "character",
gene2Symbol = "character",
geneSets = "list",
readable = "logical"
),
prototype=prototype(readable = FALSE)
)
+result变量格式
+
enrichResult中最重要的是result,是储存富集结果的dataframe。
result变量与clusterProfiler富集分析中保存ego的结果文件是一致的。
+
ego@result[c(13,14),] #查看ego的result变量的13,14行 |
一般而言result有9列。这里因为用enrichGO富集时ont参数选择ALL,结果就会在第一列前多一列ONTOLOGY。
+-
+
- 第一列是ID,也就是富集通路的编号(GO:0010222); +
- 第二列是Description,也就是富集通路的名称; +
- 第三列是GeneRatio,也就是要富集的基因中在对应通路中的比例; +
- 第4列是BgRation,也就是对应通过的基因在全基因组注释中的比例; +
- 第5,6,7列都是统计检验的结果; +
- 第8列是geneID,也就是富集到基因的名字,多个geneID是以斜线隔开的; +
- 第9列是Count,也就是富集到的基因数目。 +
1.2. 输入数据准备
根据不同情况为clusterProfiler的可视化准备输入数据。
+-
+
接着clusterProfiler富集分析做可视化
+
如果是接着clusterProfiler的enrichGO(),gseGO(),enricher(),gseGO()等函数的结果ego,不要关闭R环境,在R里直接进行用于下一步可视化即可。
+保存的clusterProfiler富集分析结果做可视化
+
+
-
+
- 如果是clusterProfiler的enrichGO(),gseGO(),enricher(),gseGO()等函数的结果
ego保存成的文件,已关闭R环境。
+ - 可导入文件,新建enrichResult对象ego,再进行下一步可视化。 +
- 这里假设用R命令
write.table(as.data.frame(ego),"go_enrich.csv",sep="\t",row.names =F,quote=F)保存ego在go_enrich.csv文件。data<-read.table("go_enrich.csv",sep="\t",header=T,quote="")
head(data,2) #查看data前2行
ONTOLOGY ID Description GeneRatio
1 BP GO:0010222 stem vascular tissue pattern formation 12/349
2 BP GO:0010588 cotyledon vascular tissue pattern formation 12/349
BgRatio pvalue p.adjust qvalue
1 29/16975 1.792157e-13 2.107577e-10 1.790270e-10
2 39/16975 1.122611e-11 6.600951e-09 5.607145e-09
geneID
1 mc11300/mc11301/mc19080/mc19081/mc26300/mc31693/mc37850/mc40780/mc40781/mc40782/mc40784/mc40918
2 mc11300/mc11301/mc19080/mc19081/mc26300/mc31693/mc37850/mc40780/mc40781/mc40782/mc40784/mc40918
Count
1 12
2 12
geneID_all <- unlist(apply(as.matrix(data$geneID),1,function(x) unlist(strsplit(x,'/')))) #得到富集到的所用geneID
ego<-new("enrichResult", result=data, gene=geneID_all, pvalueCutoff=0.01,pAdjustMethod="BH",qvalueCutoff=0.05,ontology="BP",keytype="GID",universe='Unknown',geneSets=list(),organism="Unknown",readable=FALSE) #把data内容赋值给ego的result,geneID_all赋值给gene,每个富集到的GO对应的gene集应该赋值给geneSets(数据是字典(键值对是GOID和geneIDs)组成的列表,这里直接给了空的),ontology与enrichGO分析的ont参数一致,这里的pvalueCutoff=0.01,pAdjustMethod="BH",qvalueCutoff=0.05根据富集分析参数的设置,或者随意设置或者不设置也不会影响可视化。
+
-
+
- 其他来源富集分析结果可视化
如果是其他软件的富集分析结果,可以根据ego的result变量格式进行修改格式,改成go_enrich.csv相同的格式的文件,再按照上面的步骤导入文件,并保存到新建的ego对象。即可用clusterProfiler的可视化包可视化其他软件的富集分析结果了。
+
2. 功能富集结果可视化
下面的可视化大多基于在R中已获得富集分析的结果ego。
+2.1. enrichplot包
enrichplot包有几种可视化方法来解释富集结果,支持clusterProfiler获得的ORA和GSEA富集结果。
+2.1.1. 安装和载入
安装和载入enrichplot包
+BiocManager::install("enrichplot") |
2.1.2. 可视化包
-
+
- 推荐dotplot或barplot可视化前10个GO Terms条目。 +
- 推荐goplot有向无环图查看富集的GO Terms间的关系。 +
-
+
- 可视化barplot —— 条形图
将富集分数(例如p 值)和基因计数或比率描述为条形高度和颜色。横轴为该GO term下的差异基因个数,纵轴为富集到的GO Terms的描述信息, showCategory指定展示的GO Terms的个数为20个,默认展示显著富集的top10个,即p.adjust最小的10个。
+
barplot(ego, showCategory=20, title="EnrichmentGO_MF")
使用mutate导出的其他变量也可以用作条形高度或颜色。
+mutate(ego, qscore = -log(p.adjust, base=10)) %>% |
 +
+
+
+Figure 1. Bar plot of enriched terms
from clusterProfiler book
-
+
- 可视化dotplot —— 点阵图
dotplot(edo, showCategory=30) + ggtitle("dotplot for ORA")
+
dotplot(edo2, showCategory=30) + ggtitle("dotplot for GSEA")
散点图,横坐标为GeneRatio,纵坐标为富集到的GO Terms的描述信息,showCategory指定展示的GO Terms的个数,默认展示显著富集的top10个,即p.adjust最小的10个。
+ +
+
+
+Figure 2. Dot plot of enriched terms
from clusterProfiler book
-
+
- 可视化cnetplot —— 类别网络图
cnetplot 将基因和生物学概念(例如 GO 术语或 KEGG 通路)的联系描述为一个网络(有助于查看哪些基因涉及富集通路和可能属于多个注释类别的基因)。对于基因和富集的GO terms之间的对应关系进行展示,如果一个基因位于一个GO Terms下,则将该基因与GO连线。图中灰色的点代表基因,黄色的点代表富集到的GO terms, 默认画top5富集到的GO terms, GO 节点的大小对应富集到的基因个数。
+
cnetplot(ego, categorySize = "pvalue", foldChange = gene_list
## convert gene ID to Symbol |
 +
+
+
+Figure 3. Network plot of enriched terms
from clusterProfiler book
-
+
- 可视化heatplot —— 类热图功能分类
同样使用edox。
heatplot类似cnetplot,而显示为热图的关系。
如果用户想要显示大量重要术语,那么类别网络图可能会过于复杂。在heatplot能够简化结果和更容易识别的表达模式。
+
p1 <- heatplot(edox, showCategory=5) |
 +
+
+
+Figure 4. Heatmap plot of enriched terms
from clusterProfiler book
-
+
- 可视化treeplot —— 树状图
treeplot()函数执行丰富术语的层次聚类。它依赖于pairwise_termsim()函数计算的丰富项的成对相似性,默认情况下使用 Jaccard 的相似性指数 (JC)。如果支持,用户还可以使用语义相似度值(例如,GO、DO和MeSH)。
+
默认聚合方法treeplot()是ward.D,用户可以通过hclust_method参数指定其他方法(例如,’average’、’complete’、’median’、’centroid’等。
+treeplot()函数会将树切割成几个子树(由nCluster参数指定(默认为 5))并使用高频词标记子树。
+edox2 <- pairwise_termsim(edox) |
 +
+
+
+Figure 5. Tree plot of enriched terms
from clusterProfiler book
-
+
- 可视化emapplot —— 富集图
对于富集到的GO terms之间的基因重叠关系进行展示,如果两个GO terms系的差异基因存在重叠,说明这两个节点存在overlap关系,在图中用线条连接起来。每个节点是一个富集到的GO term, 默认画top30个富集到的GO terms, 节点大小对应该GO terms下富集到的差异基因个数,节点的颜色对应p.adjust的值,从小到大,对应蓝色到红色。
+
ego2 <- pairwise_termsim(ego) |
 +
+
+
+Figure 6. Plot for results obtained from hypergeometric test and gene set enrichment analysis. default (A), cex_category=1.5 (B), layout=”kk” (C) and cex_category=1.5,layout=”kk” (D).
from clusterProfiler book
-
+
- 可视化upsetplot —— upset图
upsetplot是cnetplot可视化基因和基因集之间复杂关联的替代方法。它强调不同基因集之间的基因重叠。
+
upsetplot(ego)
 +
+
+
+Figure 7. Upsetplot for over-representation analysis.
from clusterProfiler book
-
+
- 可视化ridgeplot —— 脊线图
ridgeplot将可视化核心富集基因的表达分布为GSEA富集类别。它帮助用户解释上调/下调的途径。
+
ridgeplot(ego)
 +
+
+
+Figure 8. Ridgeplot for gene set enrichment analysis.
from clusterProfiler book
2.2. 可视化plotGOgraph/goplot —— 有向无环图
-
+
plotGOgraph(ego, firstSigNodes=10)
+
-
+
- 有向无环图(Directed acyclic graph, DAG),矩形代表富集到的top10个GO Terms,颜色从黄到红,对应p值从大到小。和topGO做富集分析的DAG图一样。 +
当enrichGO富集分析时ont参数选了ALL时,结果文件会在第一列前增加一列ONTOLOGY为子类,这时直接用于plotGOgraph画图会报错。
试了下,下面两种方案还是会报错Error in if (!ont %in% c(“BP”, “MF”, “CC”)) { :argument is of length zero。。还是尽量在enrichGO分析时就用ont=”BP”吧。
-
+
- 可以在结果文件中筛选出特定子类(比如BP)的结果行,并删除第一列ONTOLOGY后保存文件,再读进R用于plotGOgraph画图。 +
- 也可以在R内用命令
ego2<-ego%>%filter(ONTOLOGY== "BP")筛选BP子类,接着用ego3<-ego2%>%select(!ONTOLOGY)或者ego3<-ego2[,-1]删除第一列(即ONTOLOGY列),然后用plotGOgraph(ego3)作图。
+
 +
+
+
+Figure 9. DAG图
from clusterProfiler blog
-
+
goplot(ego, showCategory = 10)
+
-
+
- igraph布局方式的有向无环图 +
 +
+
+
+Figure 10. goplot的DAG图
from clusterProfiler book
2.3. 可视化 —— wordcloud
词云的方式显示结果
+install.packages("wordcloud") |
 +
+
+
+Figure 11. wordcloud词云图
from NGS Analysis ebook
3. 导出可视化结果
-
+
- Rstudio +
如果是在Rstudio中,可以直接看到绘图结果,导出需要的文件格式即可。
+-
+
- 代码导出
pdf("ego.pdf") #如果保存png,就改成png("ego.png")
ego_fig<-barplot(x) #画图函数
print(ego_fig) #画到pdf文件
dev.off() #关闭pdf画板
+
4. references
-
+
- clusterProfiler github:https://github.com/YuLab-SMU/clusterProfiler
- clusterProfiler paper:https://www.cell.com/the-innovation/fulltext/S2666-6758(21)00066-7?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS2666675821000667%3Fshowall%3Dtrue +
- clusterProfiler book:http://yulab-smu.top/biomedical-knowledge-mining-book/index.html +
- clusterProfiler manual:https://bioconductor.org/packages/devel/bioc/manuals/clusterProfiler/man/clusterProfiler.pdf +
- clusterProfiler ducumentation:https://guangchuangyu.github.io/software/clusterProfiler/documentation/ +
- 其他来源结果可视化:https://cloud.tencent.com/developer/article/1613815 +
- wordcloud:https://learn.gencore.bio.nyu.edu/rna-seq-analysis/over-representation-analysis/
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。
 ]]>
]]> +]]>
+]]>
mitogenomics简介
-
+
- 无意中发现的一个软件。 +
- 基于已有线粒体基因组序列和基因序列,通过比对获取注释tbl格式文件,所以只适用于线粒体的基因。 +
- 包括两个转化格式的脚本mitos2fasta.py,aln2tbl.py(和python2版本)。 +
软件安装
-
+
git下载
+git clone https://github.com/IMEDEA/mitogenomics
+脚本
+
+
-
+
- 因为软件是三个脚本组成,可以直接使用脚本。 +
-
+
- 依赖 +
-
+
- python3的Biopython和argparse模块。 +
- 可以用pip安装
pip install biopython argparse。
+
软件mitogenomics
脚本mitos2fasta.py
python 3 版本,用于转化基因序列为比对到线粒体基因组序列的格式。
+-
+
命令
+mitos2fasta.py -m mito.fa -g genes.fa -c Y > assembly.fa
+输入输出
+
+
-
+
- -m mito.fa:线粒体基因组序列,fasta格式。 +
- -g genes.fa:基因序列,fasta格式。可以是软件MITOS2的输出。 +
- -c Y:是否简化基因名字(genes.fa文件的序列ID)并适应aln2tbl.py,Y/N。 +
- assembly.fa:输出保存到assembly.fa,即将线粒体基因组序列和基因序列比对好的序列格式,基因没比对的位置用-代替。 +
aln2tbl.py
python 3 版本,用于转化比对到线粒体基因组序列的基因序列(即mitos2fasta.py的输出)为tbl格式。
+aln2tbl-legacy.py是aln2tbl.py的python2版本,功能一样。
+-
+
命令
+aln2tbl.py -f assembly.fa -g genes.txt -c 1 > sample.tbl
+输入输出
+
+
-
+
- -f assembly.fa:输入文件是线粒体基因组和基因序列的比对文件,mitos2fasta.py的输出。 +
- -g genes.txt:保存了基因名称的文本文件,单行,多个基因名称间逗号分隔。 +
- -c number_genetic_code:用数字指定线粒体编码方式,植物线粒体是一般的编码方式 (1)。此外还有脊椎动物vertebrate (2), 酵母菌yeast (3), 霉菌mold, 原生动物protozoan and 腔肠动物coelenterate (4), 无脊椎动物invertebrate (5), 棘皮动物echinoderm and 扁形虫flatworm (9), 海鞘类ascidian (13)。 +
+
+sample.tbl:输出到tbl格式文件。
+
+
references
++
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>mitogenomics简介
-
-
- 无意中发现的一个软件。 -
- 基于已有线粒体基因组序列和基因序列,通过比对获取注释tbl格式文件,所以只适用于线粒体的基因。 -
- 包括两个转化格式的脚本mitos2fasta.py,aln2tbl.py(和python2版本)。 -
软件安装
-
-
git下载
-git clone https://github.com/IMEDEA/mitogenomics
-脚本
-
-
-
-
- 因为软件是三个脚本组成,可以直接使用脚本。 -
-
-
- 依赖 -
-
-
- python3的Biopython和argparse模块。 -
- 可以用pip安装
pip install biopython argparse。
-
软件mitogenomics
脚本mitos2fasta.py
python 3 版本,用于转化基因序列为比对到线粒体基因组序列的格式。
--
-
命令
-mitos2fasta.py -m mito.fa -g genes.fa -c Y > assembly.fa
-输入输出
-
-
-
-
- -m mito.fa:线粒体基因组序列,fasta格式。 -
- -g genes.fa:基因序列,fasta格式。可以是软件MITOS2的输出。 -
- -c Y:是否简化基因名字(genes.fa文件的序列ID)并适应aln2tbl.py,Y/N。 -
- assembly.fa:输出保存到assembly.fa,即将线粒体基因组序列和基因序列比对好的序列格式,基因没比对的位置用-代替。 -
aln2tbl.py
python 3 版本,用于转化比对到线粒体基因组序列的基因序列(即mitos2fasta.py的输出)为tbl格式。
-aln2tbl-legacy.py是aln2tbl.py的python2版本,功能一样。
--
-
命令
-aln2tbl.py -f assembly.fa -g genes.txt -c 1 > sample.tbl
-输入输出
-
-
-
-
- -f assembly.fa:输入文件是线粒体基因组和基因序列的比对文件,mitos2fasta.py的输出。 -
- -g genes.txt:保存了基因名称的文本文件,单行,多个基因名称间逗号分隔。 -
- -c number_genetic_code:用数字指定线粒体编码方式,植物线粒体是一般的编码方式 (1)。此外还有脊椎动物vertebrate (2), 酵母菌yeast (3), 霉菌mold, 原生动物protozoan and 腔肠动物coelenterate (4), 无脊椎动物invertebrate (5), 棘皮动物echinoderm and 扁形虫flatworm (9), 海鞘类ascidian (13)。 -
-
-sample.tbl:输出到tbl格式文件。
-
-
references
--
-
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 ]]>
]]>list.files() |
3. 案例
-
-
- 蜂斗草族的系统文章用到这个方法评估系统发育树的不一致:Out of chaos: Phylogenomics of Asian Sonerileae:https://www.sciencedirect.com/science/article/pii/S1055790322001944#b0290 -
4. references
-
-
- Quartet Sampling软件github:https://github.com/fephyfofum/quartetsampling -
- paper:https://bsapubs.onlinelibrary.wiley.com/doi/full/10.1002/ajb2.1016 -
- https://github.com/ShuiyinLIU/QS_visualization -
-
-
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 ]]>
-
]]>
- 1. R包castor
R包castor是一个可以对包含超百万类群(tips)的系统发育树进行操作的程序,功能包括修剪、重新定根、计算最近共同祖先、计算tips与树根的距离、计算成对距离等等。
-系统发育信号和平均性状深度(性状保守性)的计算,离散性状的祖先状态重建和隐藏性状预测,性状进化的模拟和拟合模型,拟合和模拟多样化模型,以Newick格式树的标定时间,比较树,读取和写入树。
-安装R包castor:install.packages("castor")
2. R包castor的get_subtree_with_tips函数
R包castor的get_subtree_with_tips函数用于根据子集类群列表从一棵大树中提取子树。
-2.1. 介绍get_subtree_with_tips函数
-
-
-get_subtree_with_tips(tree,only_tips=NULL,omit_tips=NULL,collapse_monofurcations = TRUE,force_keep_root = FALSE))
-参数说明
-
-
-
-
- tree : “phylo”类的有根树。假定根是唯一的节点,没有传入的边。 -
- only_tips : 列出要保留的提示名称的字符向量,或列出要保留的提示索引的整数向量(介于1和Ntips之间)。也可以为空。在树中找不到的filename_edges_strength中列出的提示将被悄悄忽略。 -
- omit_tips : 列出要忽略的提示名称的字符向量,或列出要忽略的提示索引的整数向量(介于1和Ntips之间)。也可以为空。在树中找不到的filename_edges_strength中列出的提示将被悄悄忽略。 -
- collapse_monofurcations : 指定是否应折叠(删除)剩余单个传出边缘的节点的逻辑。此类节点的传入和传出边缘将连接到单个边缘,连接节点的父级(或更早)和子级(或更高)。在这种情况下,返回的树将具有反映连接边的边长度。 -
- force_keep_root : 逻辑值,指定是否保留根,即使filename_points_covered_by_landmarks和子树的根只剩下一个子树。如果为FALSE和filename_points_covered_by_landmarks,则可以删除根,并且它的一个后代可以成为根。 -
2.2. get_subtree_with_tips函数的使用
-
-
- 输入 -
-
-
- species.tre:物种树 -
- subtree.list:待提取的类群名称列表,每个名称一行 -
-
-
- 提取子树 -
library(treeio) |
2.3. 批量提取子树
由于get_subtree_with_tips函数只接受单棵树的phylo数据类群作为输入,如果需要从multiphylo的多棵树中统一提取子集则需要借助get_subtrees.R脚本。
--
-
- 批量提取 -
library(treeio) |
-
-
- get_subtrees.R脚本,这里设定共有2700棵树 -
for ( i in 1:2700) { |
3. 提取后
提取子树生成的species_subtree.tre文件中的枝长有时会有:NaN符号,用子树跑PhyloNetworks的时候会报错LoadError: Expected right parenthesis after left parenthesis 6 but readN是因为识别不了NaN,需要把子树中的这个符号:NaN删除。
建议用sed -i -E "s/:[0-9.Na]+//g" species_subtree.tre命令把枝长信息都删除。
3. 案例
-
+
- 蜂斗草族的系统文章用到这个方法评估系统发育树的不一致:Out of chaos: Phylogenomics of Asian Sonerileae:https://www.sciencedirect.com/science/article/pii/S1055790322001944#b0290 +
4. references
-
-
- castor包的manual:https://cran.r-project.org/web/packages/castor/castor.pdf -
- castor包的paper:https://academic.oup.com/bioinformatics/article/34/6/1053/4582279?login=true +
- Quartet Sampling软件github:https://github.com/fephyfofum/quartetsampling +
- paper:https://bsapubs.onlinelibrary.wiley.com/doi/full/10.1002/ajb2.1016 +
- https://github.com/ShuiyinLIU/QS_visualization
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。
 ]]>
]]> ]]>
]]>
1. R包castor
R包castor是一个可以对包含超百万类群(tips)的系统发育树进行操作的程序,功能包括修剪、重新定根、计算最近共同祖先、计算tips与树根的距离、计算成对距离等等。
+系统发育信号和平均性状深度(性状保守性)的计算,离散性状的祖先状态重建和隐藏性状预测,性状进化的模拟和拟合模型,拟合和模拟多样化模型,以Newick格式树的标定时间,比较树,读取和写入树。
+安装R包castor:install.packages("castor")
2. R包castor的get_subtree_with_tips函数
R包castor的get_subtree_with_tips函数用于根据子集类群列表从一棵大树中提取子树。
+2.1. 介绍get_subtree_with_tips函数
-
+
+get_subtree_with_tips(tree,only_tips=NULL,omit_tips=NULL,collapse_monofurcations = TRUE,force_keep_root = FALSE))
+参数说明
+
+
-
+
- tree : “phylo”类的有根树。假定根是唯一的节点,没有传入的边。 +
- only_tips : 列出要保留的提示名称的字符向量,或列出要保留的提示索引的整数向量(介于1和Ntips之间)。也可以为空。在树中找不到的filename_edges_strength中列出的提示将被悄悄忽略。 +
- omit_tips : 列出要忽略的提示名称的字符向量,或列出要忽略的提示索引的整数向量(介于1和Ntips之间)。也可以为空。在树中找不到的filename_edges_strength中列出的提示将被悄悄忽略。 +
- collapse_monofurcations : 指定是否应折叠(删除)剩余单个传出边缘的节点的逻辑。此类节点的传入和传出边缘将连接到单个边缘,连接节点的父级(或更早)和子级(或更高)。在这种情况下,返回的树将具有反映连接边的边长度。 +
- force_keep_root : 逻辑值,指定是否保留根,即使filename_points_covered_by_landmarks和子树的根只剩下一个子树。如果为FALSE和filename_points_covered_by_landmarks,则可以删除根,并且它的一个后代可以成为根。 +
2.2. get_subtree_with_tips函数的使用
-
+
- 输入 +
-
+
- species.tre:物种树 +
- subtree.list:待提取的类群名称列表,每个名称一行 +
-
+
- 提取子树 +
library(treeio) |
2.3. 批量提取子树
由于get_subtree_with_tips函数只接受单棵树的phylo数据类群作为输入,如果需要从multiphylo的多棵树中统一提取子集则需要借助get_subtrees.R脚本。
+-
+
- 批量提取 +
library(treeio) |
-
+
- get_subtrees.R脚本,这里设定共有2700棵树 +
for ( i in 1:2700) { |
3. 提取后
提取子树生成的species_subtree.tre文件中的枝长有时会有:NaN符号,用子树跑PhyloNetworks的时候会报错LoadError: Expected right parenthesis after left parenthesis 6 but readN是因为识别不了NaN,需要把子树中的这个符号:NaN删除。
建议用sed -i -E "s/:[0-9.Na]+//g" species_subtree.tre命令把枝长信息都删除。
4. references
-
+
- castor包的manual:https://cran.r-project.org/web/packages/castor/castor.pdf +
- castor包的paper:https://academic.oup.com/bioinformatics/article/34/6/1053/4582279?login=true +
+
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>1. 使用Rldeogram的ideogram函数画两物种的共线性图
1.1. 输入文件
-
+
- karyotype.txt +
-
+
- Chr: 染色体号 +
- Start: 起始 +
- End: 终止 +
- fill: 染色体填充色 +
- species:物种名 +
- size: 物种名字体大小 +
- color: 物种名字体颜色 +
文件示例:
+Chr Start End fill species size color |
-
+
- synteny.txt +
-
+
- Species_1:物种1染色体号 +
- Start_1,End_1:物种1染色体区域位置 +
- Species_2:物种2染色体号 +
- Start_2,End_2:物种2染色体区域位置 +
文件示例:
+Species_1 Start_1 End_1 Species_2 Start_2 End_2 fill |
1.2. 运行
install.packages('RIdeogram') #安装RIdeogram |
1.3. 结果
结果如下图:
+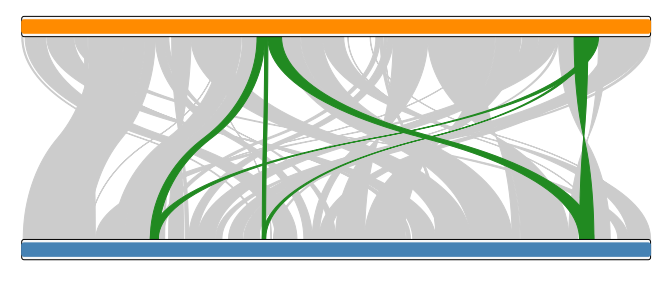 +
+
+
+Figure 1. Rldeogram绘制的同线性图
2. references
++
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>1. 转录因子(transcription factor)
转录因子(transcription factor,TF)是一种蛋白质,它通过与特定DNA序列结合来控制遗传信息从DNA到信使RNA的转录速率。
+TFs 的功能是调节——打开和关闭——基因,以确保它们在所需的细胞中在正确的时间和正确的数量表达。TF 组以协调的方式发挥作用,在整个生命过程中指导细胞分裂、细胞生长和细胞死亡;胚胎发育过程中的细胞迁移和组织;并且间歇性地响应来自细胞外的信号,例如激素。人类基因组中有多达 1600 个 TF 。转录因子是蛋白质组和调节组的成员。
+2. 植物转录因子数据库PlantTFDB
植物转录因子数据库PlantTFDB是北京大学生物信息学中心研发的数据库和网站,目前包括165个植物物种的转录因子。
+目前数据库已更新到v5.0,在网站http://planttfdb.gao-lab.org/index.php可以查看、下载和使用植物转录因子数据库。
+网站的功能包括:
+-
+
- 上传核酸或蛋白质的fasta序列,在线做转录因子的注释。 +
- 上传核酸或蛋白质的fasta序列,在线与数据库做blastx或blastp比对。 +
- 下载特定植物的TF列表,CDS或蛋白质序列。 +
- 查询特定TF和TF家族的功能描述。 +
3. 转录因子相关分析
转录因子分析可以应用的场景很多,这里介绍全基因组复制事件(WGD)后转录因子保留的分析。
+3.1. WGD后保留TF的分析
3.1.1. 思路
除了直接看WGD后保留的基因中包含了什么种类和多少数量的TF外,还可以通过利用转录因子数据库PlantTFDB来做WGD后保留的每种TF的保留模式的进一步分析。
+-
+
- 参考 +
-
+
- paper: https://www.sciencedirect.com/science/article/pii/S1674205219303594 的 Retention Analysis of Transcription Factors部分。 +
- 在博客鉴定全基因复制事件(WGD)后保留的复制基因 https://yanzhongsino.github.io/2022/10/18/bioinfo_WGD_geneRetention/ 的基础上完成WGD后保留TF的分析 +
-
+
- 基本思路 +
-
+
- 从PlantTFDB数据库下载已有物种(比如拟南芥)的TF家族,用下载的TF家族注释orthogroups。对每个WGD事件,确认每个TF家族的保留的orthogroups的数量。 +
- 有些TF家族可能会被分到几个orthogroups,为了消除一个TF家族的orthogroups的大小不均的影响,文章通过标准化计算一个保留参数R值(retention value),R值用来反映WGD事件后每个TF的保留模式。 +
-
+
- R值的计算公式:$$Rvalue=(Rs⁄Ts)/(Ra⁄Ta)=RsTa/TsRa$$,其中: +
-
+
- Rs: Number of orthogroups with retention in specific TF +
- Ts: Total number of orthogroups in specific TF +
- Ra: Number of all TF orthogroups with retention +
- Ta: Total number of TF orthogroups +
- Rs/Ts: 代表在WGD后特定TF家族保留的可能性 +
- Ra/Ta: 代表在WGD后所有TF家族保留的可能性 +
- Rvalue: 用Rs/Ts比上Ra/Ta,代表相较TF家族平均水平,特定TF家族保留的可能性的高低。Rvalue越大,特定TF家族的保留率越高。 +
3.1.2. 准备文件
-
+
- Orthogroups.txt +
-
+
- Orthofinder的结果文件/path/to/OrthoFinder/Results_xx/Orthogroups/Orthogroups.txt +
- Orthofinder运行时需要包含了下载TF的物种 +
-
+
- dup_wgd.og +
-
+
- dup_wgd.og包含了前期分析的基因复制的、涉及特定WGD保留的那些orthogroups的ID列表。 +
- 可以从WGD后保留基因的分析的结果文件
N5_filter_OG_dup.tsv中提取第二列来获取:cat N5_filter_OG_dup.tsv|cut -f2 >dup_wgd.og。
+
-
+
- 下载Ath_TF_list.txt并转化成Ath_TF_list.og +
cat Ath_TF_list.txt|sed '1d'|cut -f2 >ath_2.tem # 提取第二列geneID |
3.1.3. 统计R相关参数
-
+
- 这里的Ta、Ra、Ts、Rs可以用两种数量来代表,一种是统计TF_ID的数量,另一种是统计Orthogroups的数量。 +
-
+
- 下面的是统计TF_ID的数量,如果想要统计Orthogroups的数量,则需要在每一个值统计命令
wc -l前面加上cut -f5|sort|uniq|来提取Orthogroups并去重。
+
-
+
- 对每一个ath.tf里的Family,统计Ta,Ra,Rs和Ts值 +
for i in $(cat ath.tf); |
-
+
- 有了Ta,Ra,Rs和Ts值,接下来就可以计算Rvalue=(Rs⁄Ts)/(Ra⁄Ta)了。 +
-
+
cat ath_r.tem|sed "s/ /\t/g"|awk -F"\t" '{print $0,($1*$4)/($2*$3)}'|sed '1i\TF\tRs\tTs\tRa\tTa\tRvalue' >ath_r.txt待检查是否有效
+
3.1.4. 绘制热图
热图绘制可以参考博客https://yanzhongsino.github.io/2022/11/06/R_plot_heatmap
+-
+
- 如果只有一次WGD的TF保留结果,可以直接根据Rvalue判断哪些TF家族保留率高。 +
- 如果有多次WGD的TF保留结果,或者做了多个物种的TF数据库保留结果,可以绘制热图相互比较。 +
用R包pheatmap绘制热图,简单快捷。(notes: 画热图这里的代码还需根据数据格式调整)
+df<-read.table("tf_rvalue.txt",sep= " ", header = T,row.names = 1) |
4. references
-
+
- wiki:transcription factor: https://en.wikipedia.org/wiki/Transcription_factor +
- PlantTFDB: http://planttfdb.gao-lab.org/index.php +
- paper: https://www.sciencedirect.com/science/article/pii/S1674205219303594 +
+
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>1. 使用Rldeogram的ideogram函数画两物种的共线性图
1.1. 输入文件
-
-
- karyotype.txt -
-
-
- Chr: 染色体号 -
- Start: 起始 -
- End: 终止 -
- fill: 染色体填充色 -
- species:物种名 -
- size: 物种名字体大小 -
- color: 物种名字体颜色 -
文件示例:
-Chr Start End fill species size color |
-
-
- synteny.txt -
-
-
- Species_1:物种1染色体号 -
- Start_1,End_1:物种1染色体区域位置 -
- Species_2:物种2染色体号 -
- Start_2,End_2:物种2染色体区域位置 -
文件示例:
-Species_1 Start_1 End_1 Species_2 Start_2 End_2 fill |
1.2. 运行
install.packages('RIdeogram') #安装RIdeogram |
1.3. 结果
结果如下图:
- -
-
-
-Figure 1. Rldeogram绘制的同线性图
2. references
--
-
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 ]]>
]]>1. 转录因子(transcription factor)
转录因子(transcription factor,TF)是一种蛋白质,它通过与特定DNA序列结合来控制遗传信息从DNA到信使RNA的转录速率。
-TFs 的功能是调节——打开和关闭——基因,以确保它们在所需的细胞中在正确的时间和正确的数量表达。TF 组以协调的方式发挥作用,在整个生命过程中指导细胞分裂、细胞生长和细胞死亡;胚胎发育过程中的细胞迁移和组织;并且间歇性地响应来自细胞外的信号,例如激素。人类基因组中有多达 1600 个 TF 。转录因子是蛋白质组和调节组的成员。
-2. 植物转录因子数据库PlantTFDB
植物转录因子数据库PlantTFDB是北京大学生物信息学中心研发的数据库和网站,目前包括165个植物物种的转录因子。
-目前数据库已更新到v5.0,在网站http://planttfdb.gao-lab.org/index.php可以查看、下载和使用植物转录因子数据库。
-网站的功能包括:
--
-
- 上传核酸或蛋白质的fasta序列,在线做转录因子的注释。 -
- 上传核酸或蛋白质的fasta序列,在线与数据库做blastx或blastp比对。 -
- 下载特定植物的TF列表,CDS或蛋白质序列。 -
- 查询特定TF和TF家族的功能描述。 -
3. 转录因子相关分析
转录因子分析可以应用的场景很多,这里介绍全基因组复制事件(WGD)后转录因子保留的分析。
-3.1. WGD后保留TF的分析
3.1.1. 思路
除了直接看WGD后保留的基因中包含了什么种类和多少数量的TF外,还可以通过利用转录因子数据库PlantTFDB来做WGD后保留的每种TF的保留模式的进一步分析。
--
-
- 参考 -
-
-
- paper: https://www.sciencedirect.com/science/article/pii/S1674205219303594 的 Retention Analysis of Transcription Factors部分。 -
- 在博客鉴定全基因复制事件(WGD)后保留的复制基因 https://yanzhongsino.github.io/2022/10/18/bioinfo_WGD_geneRetention/ 的基础上完成WGD后保留TF的分析 -
-
-
- 基本思路 -
-
-
- 从PlantTFDB数据库下载已有物种(比如拟南芥)的TF家族,用下载的TF家族注释orthogroups。对每个WGD事件,确认每个TF家族的保留的orthogroups的数量。 -
- 有些TF家族可能会被分到几个orthogroups,为了消除一个TF家族的orthogroups的大小不均的影响,文章通过标准化计算一个保留参数R值(retention value),R值用来反映WGD事件后每个TF的保留模式。 -
-
-
- R值的计算公式:$$Rvalue=(Rs⁄Ts)/(Ra⁄Ta)=RsTa/TsRa$$,其中: -
-
-
- Rs: Number of orthogroups with retention in specific TF -
- Ts: Total number of orthogroups in specific TF -
- Ra: Number of all TF orthogroups with retention -
- Ta: Total number of TF orthogroups -
- Rs/Ts: 代表在WGD后特定TF家族保留的可能性 -
- Ra/Ta: 代表在WGD后所有TF家族保留的可能性 -
- Rvalue: 用Rs/Ts比上Ra/Ta,代表相较TF家族平均水平,特定TF家族保留的可能性的高低。Rvalue越大,特定TF家族的保留率越高。 -
3.1.2. 准备文件
-
-
- Orthogroups.txt -
-
-
- Orthofinder的结果文件/path/to/OrthoFinder/Results_xx/Orthogroups/Orthogroups.txt -
- Orthofinder运行时需要包含了下载TF的物种 -
-
-
- dup_wgd.og -
-
-
- dup_wgd.og包含了前期分析的基因复制的、涉及特定WGD保留的那些orthogroups的ID列表。 -
- 可以从WGD后保留基因的分析的结果文件
N5_filter_OG_dup.tsv中提取第二列来获取:cat N5_filter_OG_dup.tsv|cut -f2 >dup_wgd.og。
-
-
-
- 下载Ath_TF_list.txt并转化成Ath_TF_list.og -
cat Ath_TF_list.txt|sed '1d'|cut -f2 >ath_2.tem # 提取第二列geneID |
3.1.3. 统计R相关参数
-
-
- 这里的Ta、Ra、Ts、Rs可以用两种数量来代表,一种是统计TF_ID的数量,另一种是统计Orthogroups的数量。 -
-
-
- 下面的是统计TF_ID的数量,如果想要统计Orthogroups的数量,则需要在每一个值统计命令
wc -l前面加上cut -f5|sort|uniq|来提取Orthogroups并去重。
-
-
-
- 对每一个ath.tf里的Family,统计Ta,Ra,Rs和Ts值 -
for i in $(cat ath.tf); |
-
-
- 有了Ta,Ra,Rs和Ts值,接下来就可以计算Rvalue=(Rs⁄Ts)/(Ra⁄Ta)了。 -
-
-
cat ath_r.tem|sed "s/ /\t/g"|awk -F"\t" '{print $0,($1*$4)/($2*$3)}'|sed '1i\TF\tRs\tTs\tRa\tTa\tRvalue' >ath_r.txt待检查是否有效
-
3.1.4. 绘制热图
热图绘制可以参考博客https://yanzhongsino.github.io/2022/11/06/R_plot_heatmap
--
-
- 如果只有一次WGD的TF保留结果,可以直接根据Rvalue判断哪些TF家族保留率高。 -
- 如果有多次WGD的TF保留结果,或者做了多个物种的TF数据库保留结果,可以绘制热图相互比较。 -
用R包pheatmap绘制热图,简单快捷。(notes: 画热图这里的代码还需根据数据格式调整)
-df<-read.table("tf_rvalue.txt",sep= " ", header = T,row.names = 1) |
4. references
-
-
- wiki:transcription factor: https://en.wikipedia.org/wiki/Transcription_factor -
- PlantTFDB: http://planttfdb.gao-lab.org/index.php -
- paper: https://www.sciencedirect.com/science/article/pii/S1674205219303594 -
-
-
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 ]]>
]]>在根据博客hexo建站,github.io发布,多终端同步配置了hexo网站(使用next主题)的基础上,记录了博客日常撰写、备份。
-1. blog的categories和tags
categories和tags的记录
---- |
2. 日常blog撰写和备份操作
在做好blog搭建后,blog撰写和日常管理可参考这部分内容。
-2.1. blog同步
养成习惯,每次开始撰写blog前都通过git bash进入工作区,进行git pull命令把github端的hexo分支的更新(更新可能是其他终端上提交的)同步到本地,实现多终端的内容完全同步。
但如果本地有未提交的更新,则千万不要用git pull,否则会覆盖本地更新;直接进入下一步;直到使用git add .,git commit -m "submit",git push origin hexo提交备份本地更新到github端的hexo分支后才可以使用git pull(一般是在其他终端,把github的hexo分支更新拉到其他终端设备使用)。
2.2. blog撰写
在本地source/_posts下添加和修改md文档实现blog的日常撰写和修改。
-使用命令hexo new "newpostname"可以在hexo/source/_posts下新建一个newpostname.md的文件,这个文件以scaffolds/post.md为模板,修改scaffolds/post.md文件可以修改hexo new命令生成的新blog文件样式。
2.3. blog备份
只要blog有更改或者新增,或者配置文件有修改,即工作区(即本地的hexo目录或github.io目录)有文件修改,则建议对文件进行备份到GitHub端的hexo分支。
用三条命令git add .,git commit -m "submit",git push origin hexo备份工作区,包括md博客源文件和hexo部署到github端的hexo分支。三条命令执行前建议通过hexo clean清除缓存和public目录,以免备份不需要的文件。
2.4. blog发布
可根据自身需求决定是否发布blog到github.io网站,一般写的blog完整程度比较高时可以发布。使用hexo clean & hexo g -d命令,根据source/_posts下的博客源文件生成public目录(网站html并同步到github端的master分支,即发布blog到github.io网站。
总结一下,在配置好写作环境后的任意一台终端的日常工作流应该是:
--
-
git pull同步远程github库的hexo更新到本地
-hexo new "newblog"在source/_posts/下添加md格式的blog,或者修改已有的blog
-git add .,git commit -m "commit notes",git push把修改备份到github端
-- 下次写作重复以上三个步骤 -
- 直至blog完善成熟后,用命令
hexo clean & hexo g -d生成网站并部署到github.io
-
-
-
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 ]]>
]]>在根据博客hexo建站,github.io发布,多终端同步配置了hexo网站(使用next主题)的基础上,记录了博客日常撰写、备份。
+1. blog的categories和tags
categories和tags的记录
+--- |
2. 日常blog撰写和备份操作
在做好blog搭建后,blog撰写和日常管理可参考这部分内容。
+2.1. blog同步
养成习惯,每次开始撰写blog前都通过git bash进入工作区,进行git pull命令把github端的hexo分支的更新(更新可能是其他终端上提交的)同步到本地,实现多终端的内容完全同步。
但如果本地有未提交的更新,则千万不要用git pull,否则会覆盖本地更新;直接进入下一步;直到使用git add .,git commit -m "submit",git push origin hexo提交备份本地更新到github端的hexo分支后才可以使用git pull(一般是在其他终端,把github的hexo分支更新拉到其他终端设备使用)。
2.2. blog撰写
在本地source/_posts下添加和修改md文档实现blog的日常撰写和修改。
+使用命令hexo new "newpostname"可以在hexo/source/_posts下新建一个newpostname.md的文件,这个文件以scaffolds/post.md为模板,修改scaffolds/post.md文件可以修改hexo new命令生成的新blog文件样式。
2.3. blog备份
只要blog有更改或者新增,或者配置文件有修改,即工作区(即本地的hexo目录或github.io目录)有文件修改,则建议对文件进行备份到GitHub端的hexo分支。
用三条命令git add .,git commit -m "submit",git push origin hexo备份工作区,包括md博客源文件和hexo部署到github端的hexo分支。三条命令执行前建议通过hexo clean清除缓存和public目录,以免备份不需要的文件。
2.4. blog发布
可根据自身需求决定是否发布blog到github.io网站,一般写的blog完整程度比较高时可以发布。使用hexo clean & hexo g -d命令,根据source/_posts下的博客源文件生成public目录(网站html并同步到github端的master分支,即发布blog到github.io网站。
总结一下,在配置好写作环境后的任意一台终端的日常工作流应该是:
+-
+
git pull同步远程github库的hexo更新到本地
+hexo new "newblog"在source/_posts/下添加md格式的blog,或者修改已有的blog
+git add .,git commit -m "commit notes",git push把修改备份到github端
+- 下次写作重复以上三个步骤 +
- 直至blog完善成熟后,用命令
hexo clean & hexo g -d生成网站并部署到github.io
+
+
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>1. ncRNA
非编码RNA(Non-coding RNA, ncRNA) 包括rRNA,tRNA,snRNA,snoRNA 和microRNA 等不编码蛋白质的RNA,它们转录后直接在RNA 水平上就能行使各自的生物学功能,并不需要翻译成蛋白质。
-2. 注释软件
-
-
- 非编码RNA种类繁多,且结构特征各不相同,所以开发出了许多注释特定某一类RNA的软件,比如tRNAScan-SE预测tRNA,rnammer预测rRNA,snoScan 搜索带C/D盒的snoRNAs,SnoGps 搜索带H/ACA盒的snoRNAs,mirScan搜索microRNA等。 -
- Sanger实验室开发了Infernal软件,建立了1600多个RNA家族,并对每个家族建立了一致性二级结构和协方差模型,形成了Rfam数据库。采用Rfam数据库中的每个RNA的协方差模型,结合Infernal软件可以预测出已有RNA家族的新成员,只是特异性比较差。 -
如果不是专门研究ncRNA,可以用Infernal注释所有ncRNA。如果需要更精细的注释,则可以选择特定软件注释特定RNA。
-这篇博客是介绍用Infernal程序与Rfam数据库一起用来注释与数据库中已知ncRNA同源的序列(这里用来注释完整的基因组)。注释结果包括tRNA,rRNA,snRNA,snoRNA和miRNA等。
-3. Infernal
Infernal全称是”INFERence of RNA ALignment”,是一个用来检索DNA序列数据库中RNA序列和结构相似性的软件,通过协方差模型covariance models (CMs)来实现。
-3.1. 安装Infernal
conda install -c bioconda infernal
现在安装的是v1.1.4
安装后可使用的命令包括:
--
-
- cmpress:对cm文件进行压缩并建立索引。 -
- cmscan:用提交的序列在cm数据库中进行检索。 -
- cmalign:将RNA序列同协方差模型进行比对,并输出为stockholm格式。 -
- cmbuild:通过多序列比对结果建立一个协方差模型,并保存在新文件中。 -
- cmcalibrate:对协方差模型(CM)进行校准,在使用cmsearch和cmscan前CM模型需要先经过这个程序的处理。 -
- cmconvert:用于格式转换,是将infernal 1.0以后的CM转化为当前版本需要的CM。但对1.0之前的版本无效。 -
- cmemit:一个采样程序,从CM中进行采样并输出。 -
- cmfetch:从一个大的CM文件中获取一个或多个CM。 -
- cmsearch:用提交的CM模型在序列数据库中进行检索。 -
- cmstat:对CM文件中的CM模型进行统计汇总。 -
4. Rfam
Rfam是RNA family数据库,包括ncRNA序列和ncRNA的二级结构,每个family用多序列比对和协方差模型covariance model (CM)来表示。
--
-
- 下载Rfam数据库 -
-
-
wget http://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.cm.gz
在Rfam网站下载Rfam最新版本的CM数据库(目前是Rfam 14.7)
-gunzip Rfam.cm.gz
解压数据库
-
-
-
- 下载clanin -
-
-
wget http://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.clanin
在Rfam网站下载Rfam数据库配套的clanin文件。
-
5. 注释ncRNA
5.1. 建库
-
-
cmpress Rfam.cm
使用cmpress压缩并建立索引,生成Rfam.cm.i1f, Rfam.cm.i1i, Rfam.cm.i1m, Rfam.cm.i1p。
-
5.2. 注释
-
-
- 序列索引
推荐的参数:
-
nohup cmscan -Z 512 --cut_ga --rfam --nohmmonly --fmt 2 --tblout sample.tblout -o sample.result --clanin Rfam.clanin Rfam.cm genome.fa &
-
-
- -Z:根据基因组大小来定,基因组大小的2倍,Mb单位,选一个整数。比如256Mb的基因组,-Z 512。 -
--cut_ga --rfam --nohmmonly --fmt 2:推荐使用
-- –tblout sample.tblout:指定table格式输出文件 -
- -o sample.result:指定比对结果输出文件 -
- –clanin Rfam.clanin:指定clanin文件 -
- Rfam.cm genome.fa:指定数据库Rfam.cm和基因组genome.fa -
note:-o sample.result要放在Rfam.cm genome.fa前面,否则报错。
-此步骤耗时参考:250Mb基因组,默认线程,耗时2.5h。
--
-
- 结果文件 -
-
-
- sample.result:比对结果 -
- sample.tblout:table格式结果 -
5.3. 整理结果
5.3.1. 将注释结果整理成gff3文件
gff3文件可用于提交注释到数据库。
-用perl脚本infernal-tblout2gff.pl实现,脚本来自https://www.cnblogs.com/jessepeng/p/15392809.html。
-perl infernal-tblout2gff.pl --cmscan --fmt2 sample.tblout >sample.infernal.ncRNA.gff3
5.3.2. 统计各类ncRNA总数
-
-
整理注释结果文件sample.tblout
-
提取必需的列,非重叠区域或重叠区域得分高的区域awk 'BEGIN{OFS="\t";}{if(FNR==1) print "target_name\taccession\tquery_name\tquery_start\tquery_end\tstrand\tscore\tEvalue"; if(FNR>2 && $20!="=" && $0!~/^#/) print $2,$3,$4,$10,$11,$12,$17,$18; }' sample.tblout >sample.tblout.xls
-下载rfam注释
-
-
-
-
- 在rfam官网,选择【SEARCH】-【Entry type】 -
- 然后选中所有的Entry types(包括Gene,Intron,Cis-regulatory element),点击【Submit】,会列出所有RNA family的注释信息。 -
- 手动选择所有注释信息,复制,粘贴到新建的空白文本文件rfam.txt并保存。 -
- 把rfam.txt传输到服务器,最好用
dos2unix rfam.txt转换文件格式为unix版本。
- - 拆分第三列
cat rfam.txt | awk 'BEGIN {FS=OFS="\t"}{split($3,x,";");class=x[2];print $1,$2,$3,$4,class}' > rfam_anno.txt
-
rfam注释文件rfam_anno.txt包含了所有rfam的类型type和功能描述description信息。
--
-
- 统计ncRNA注释结果
awk 'BEGIN{OFS=FS="\t"}ARGIND==1{a[$2]=$5;}ARGIND==2{type=a[$1]; if(type=="") type="Others"; count[type]+=1;}END{for(type in count) print type, count[type];}' rfam_anno.txt sample.tblout.xls >sample.ncRNA.statistic
-
sample.ncRNA.statistic输出示例:
-riboswitch 1 |
-
-
- 统计细分分类 -
-
-
- 也可以根据细分分类分别统计,细分分类参考rfam官网,【SEARCH】-【Entry type】。 -
- 可参考的统计值包括每个细分ncRNA的数量(copy),平均长度(average length),总长(total length),总长占基因组的比例(Percent of the genome)等 -
- 统计后整理成发表文章用的表格。 -
比如:snRNA包括snoRNA和splicing,snoRNA包括CD-box,HACA-box和scaRNA。下面用统计CD-box这个细分分类的ncRNA举例。
--
-
- 提取CD-box的Accession(RF00000格式):
grep "CD-box" rfam_anno.txt |cut -f1 >cdbox.tem
- - 提取注释到的CD-box信息:
grep -f cdbox.tem mc.tblout.xls >cdbox.txt
- - cdbox.txt的行数就是CD-box的数量;利用第四五列的位置信息,即可统计平均长度(average length),总长(total length),注意正反链。 -
6. references
-
-
- https://www.cnblogs.com/jessepeng/p/15392809.html -
- http://www.360doc.com/content/18/1119/05/52645714_795799901.shtml -
- https://genehub.wordpress.com/2019/08/08/%E6%A4%8D%E7%89%A9%E5%9F%BA%E5%9B%A0%E7%BB%84ncrna%E9%A2%84%E6%B5%8B%EF%BC%88trna%E3%80%81rrna%E3%80%81snrna%E3%80%81mirna%EF%BC%89/ -
- http://embracethesky.cn/2018/07/08/%e4%bd%bf%e7%94%a8infernal%e5%af%b9rfam-12%e8%bf%9b%e8%a1%8crna%e6%b3%a8%e9%87%8a/#more-99 -
-
-
-
- 欢迎关注微信公众号:生信技工 -
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 -
 ]]>
]]>1. ncRNA
非编码RNA(Non-coding RNA, ncRNA) 包括rRNA,tRNA,snRNA,snoRNA 和microRNA 等不编码蛋白质的RNA,它们转录后直接在RNA 水平上就能行使各自的生物学功能,并不需要翻译成蛋白质。
+2. 注释软件
-
+
- 非编码RNA种类繁多,且结构特征各不相同,所以开发出了许多注释特定某一类RNA的软件,比如tRNAScan-SE预测tRNA,rnammer预测rRNA,snoScan 搜索带C/D盒的snoRNAs,SnoGps 搜索带H/ACA盒的snoRNAs,mirScan搜索microRNA等。 +
- Sanger实验室开发了Infernal软件,建立了1600多个RNA家族,并对每个家族建立了一致性二级结构和协方差模型,形成了Rfam数据库。采用Rfam数据库中的每个RNA的协方差模型,结合Infernal软件可以预测出已有RNA家族的新成员,只是特异性比较差。 +
如果不是专门研究ncRNA,可以用Infernal注释所有ncRNA。如果需要更精细的注释,则可以选择特定软件注释特定RNA。
+这篇博客是介绍用Infernal程序与Rfam数据库一起用来注释与数据库中已知ncRNA同源的序列(这里用来注释完整的基因组)。注释结果包括tRNA,rRNA,snRNA,snoRNA和miRNA等。
+3. Infernal
Infernal全称是”INFERence of RNA ALignment”,是一个用来检索DNA序列数据库中RNA序列和结构相似性的软件,通过协方差模型covariance models (CMs)来实现。
+3.1. 安装Infernal
conda install -c bioconda infernal
现在安装的是v1.1.4
安装后可使用的命令包括:
+-
+
- cmpress:对cm文件进行压缩并建立索引。 +
- cmscan:用提交的序列在cm数据库中进行检索。 +
- cmalign:将RNA序列同协方差模型进行比对,并输出为stockholm格式。 +
- cmbuild:通过多序列比对结果建立一个协方差模型,并保存在新文件中。 +
- cmcalibrate:对协方差模型(CM)进行校准,在使用cmsearch和cmscan前CM模型需要先经过这个程序的处理。 +
- cmconvert:用于格式转换,是将infernal 1.0以后的CM转化为当前版本需要的CM。但对1.0之前的版本无效。 +
- cmemit:一个采样程序,从CM中进行采样并输出。 +
- cmfetch:从一个大的CM文件中获取一个或多个CM。 +
- cmsearch:用提交的CM模型在序列数据库中进行检索。 +
- cmstat:对CM文件中的CM模型进行统计汇总。 +
4. Rfam
Rfam是RNA family数据库,包括ncRNA序列和ncRNA的二级结构,每个family用多序列比对和协方差模型covariance model (CM)来表示。
+-
+
- 下载Rfam数据库 +
-
+
wget http://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.cm.gz
在Rfam网站下载Rfam最新版本的CM数据库(目前是Rfam 14.7)
+gunzip Rfam.cm.gz
解压数据库
+
-
+
- 下载clanin +
-
+
wget http://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.clanin
在Rfam网站下载Rfam数据库配套的clanin文件。
+
5. 注释ncRNA
5.1. 建库
-
+
cmpress Rfam.cm
使用cmpress压缩并建立索引,生成Rfam.cm.i1f, Rfam.cm.i1i, Rfam.cm.i1m, Rfam.cm.i1p。
+
5.2. 注释
-
+
- 序列索引
推荐的参数:
+
nohup cmscan -Z 512 --cut_ga --rfam --nohmmonly --fmt 2 --tblout sample.tblout -o sample.result --clanin Rfam.clanin Rfam.cm genome.fa &
-
+
- -Z:根据基因组大小来定,基因组大小的2倍,Mb单位,选一个整数。比如256Mb的基因组,-Z 512。 +
--cut_ga --rfam --nohmmonly --fmt 2:推荐使用
+- –tblout sample.tblout:指定table格式输出文件 +
- -o sample.result:指定比对结果输出文件 +
- –clanin Rfam.clanin:指定clanin文件 +
- Rfam.cm genome.fa:指定数据库Rfam.cm和基因组genome.fa +
note:-o sample.result要放在Rfam.cm genome.fa前面,否则报错。
+此步骤耗时参考:250Mb基因组,默认线程,耗时2.5h。
+-
+
- 结果文件 +
-
+
- sample.result:比对结果 +
- sample.tblout:table格式结果 +
5.3. 整理结果
5.3.1. 将注释结果整理成gff3文件
gff3文件可用于提交注释到数据库。
+用perl脚本infernal-tblout2gff.pl实现,脚本来自https://www.cnblogs.com/jessepeng/p/15392809.html。
+perl infernal-tblout2gff.pl --cmscan --fmt2 sample.tblout >sample.infernal.ncRNA.gff3
5.3.2. 统计各类ncRNA总数
-
+
整理注释结果文件sample.tblout
+
提取必需的列,非重叠区域或重叠区域得分高的区域awk 'BEGIN{OFS="\t";}{if(FNR==1) print "target_name\taccession\tquery_name\tquery_start\tquery_end\tstrand\tscore\tEvalue"; if(FNR>2 && $20!="=" && $0!~/^#/) print $2,$3,$4,$10,$11,$12,$17,$18; }' sample.tblout >sample.tblout.xls
+下载rfam注释
+
+
-
+
- 在rfam官网,选择【SEARCH】-【Entry type】 +
- 然后选中所有的Entry types(包括Gene,Intron,Cis-regulatory element),点击【Submit】,会列出所有RNA family的注释信息。 +
- 手动选择所有注释信息,复制,粘贴到新建的空白文本文件rfam.txt并保存。 +
- 把rfam.txt传输到服务器,最好用
dos2unix rfam.txt转换文件格式为unix版本。
+ - 拆分第三列
cat rfam.txt | awk 'BEGIN {FS=OFS="\t"}{split($3,x,";");class=x[2];print $1,$2,$3,$4,class}' > rfam_anno.txt
+
rfam注释文件rfam_anno.txt包含了所有rfam的类型type和功能描述description信息。
+-
+
- 统计ncRNA注释结果
awk 'BEGIN{OFS=FS="\t"}ARGIND==1{a[$2]=$5;}ARGIND==2{type=a[$1]; if(type=="") type="Others"; count[type]+=1;}END{for(type in count) print type, count[type];}' rfam_anno.txt sample.tblout.xls >sample.ncRNA.statistic
+
sample.ncRNA.statistic输出示例:
+riboswitch 1 |
-
+
- 统计细分分类 +
-
+
- 也可以根据细分分类分别统计,细分分类参考rfam官网,【SEARCH】-【Entry type】。 +
- 可参考的统计值包括每个细分ncRNA的数量(copy),平均长度(average length),总长(total length),总长占基因组的比例(Percent of the genome)等 +
- 统计后整理成发表文章用的表格。 +
比如:snRNA包括snoRNA和splicing,snoRNA包括CD-box,HACA-box和scaRNA。下面用统计CD-box这个细分分类的ncRNA举例。
+-
+
- 提取CD-box的Accession(RF00000格式):
grep "CD-box" rfam_anno.txt |cut -f1 >cdbox.tem
+ - 提取注释到的CD-box信息:
grep -f cdbox.tem mc.tblout.xls >cdbox.txt
+ - cdbox.txt的行数就是CD-box的数量;利用第四五列的位置信息,即可统计平均长度(average length),总长(total length),注意正反链。 +
6. references
-
+
- https://www.cnblogs.com/jessepeng/p/15392809.html +
- http://www.360doc.com/content/18/1119/05/52645714_795799901.shtml +
- https://genehub.wordpress.com/2019/08/08/%E6%A4%8D%E7%89%A9%E5%9F%BA%E5%9B%A0%E7%BB%84ncrna%E9%A2%84%E6%B5%8B%EF%BC%88trna%E3%80%81rrna%E3%80%81snrna%E3%80%81mirna%EF%BC%89/ +
- http://embracethesky.cn/2018/07/08/%e4%bd%bf%e7%94%a8infernal%e5%af%b9rfam-12%e8%bf%9b%e8%a1%8crna%e6%b3%a8%e9%87%8a/#more-99 +
+
-
+
- 欢迎关注微信公众号:生信技工 +
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。 +
 ]]>
]]>- 表示蛋白质长度与预测的蛋白质长度不匹配,运行错误 -
- 建议重跑table2asn,报错持续存在就写邮件把sample.sqn和运行的命令行发给NCBI(genomes@ncbi.nlm.nih.gov)让帮忙修改这个错误。 +
- 建议重跑table2asn,报错持续存在就写邮件把sample.sqn和运行的命令行发给NCBI(genomes@ncbi.nlm.nih.gov)让帮忙修改这个错误。
-
diff --git a/tags/HiSat2/index.html b/tags/HiSat2/index.html
index 7ee3a5f63a..76ba31495e 100644
--- a/tags/HiSat2/index.html
+++ b/tags/HiSat2/index.html
@@ -23,7 +23,7 @@
-
+
@@ -33,7 +33,7 @@
-
+
-